高、低剩余采食量滩羊瘤胃微生物的差异
English
-
饲料成本占养殖总成本的65%~70%,增加养殖经济效益,提高饲料效率是关键。剩余采食量(residual feed intake, RFI)为基于动物体况和生产性能的实际饲料摄入量和其预期饲料摄入量之间的差异[1],被广泛应用于评估各种家畜动物饲料效率,RFI较低的个体饲料效率更高。然而RFI表型受许多因素影响,如日粮、喂养方式(如喂养的持续时间和次数)、瘤胃发酵、体况状况(脂肪和蛋白质)、代谢过程、运动和饲喂时所产生的热增耗[2]。目前鲜有研究报道性别与饲料效率间的差异关系,仅Ahmed等[3]发现性别影响不同RFI肉牛瘤胃上皮基因差异表达,对生长性能和瘤胃细菌没有显著影响。
瘤胃是瘤胃微生物的主要发酵场所,短链脂肪酸是瘤胃发酵的最终产物,是反刍动物能量的主要供应来源。因此,瘤胃功能与饲料效率密切相关。瘤胃发酵中会产生一些不必要的能量损失。甲烷是造成温室效应的主要气体之一,反刍动物瘤胃产生的CH4约7.7 × 107 t,绝大部分CH4气体来源于反刍动物。据报道,反刍动物胃肠道CH4消耗占总能量摄入的5%~10%[4]。肉牛甲烷的日产率与RFI正相关,低RFI个体甲烷排放量少。因此,低RFI动物个体的选育不仅可以降低动物的养殖成本,还可以减小养殖对环境的污染。Guan等[5]报道了瘤胃微生物群及其发酵参数与牛的饲料效率之间的潜在联系,低RFI较高RFI个体拥有特异性菌群。Deborah等[6]通过16S rRNA测序技术评价内洛尔肉牛瘤胃微生物群与饲料效率的关系发现,低RFI和高RFI牛的细菌组成差异主要与Lachnospiraceae、Ruminococcaceae、Christensenellaceae以及Prevotella的成员有关。对不同RFI绵羊的16s RNA测序发现,低RFI绵羊表现出更丰富和更多样化的微生物菌群[7]。瘤胃微生物代谢网络异常也与饲料效率密切相关[8]。对不同RFI肉牛瘤胃微生物18s rRNA测序发现,原虫群落的未分配类群和系统发育多样性的差异可能是造成肉牛饲料效率表型差异的原因[9]。Liang等[10]发现,低RFI动物拥有更低的pH,并且低RFI瘤胃环境比高RFI更稳定,有利于饲料发酵。目前研究表明瘤胃菌群与饲料效率密切相关。因此,本研究选择大群体公母滩羊,对其进行不限定运动饲养,探究不同RFI公母滩羊瘤胃微生物差异,确定影响饲料效率的关键微生物,为滩羊选育实践提供理论支持。
1. 材料与方法
1.1 试验羊饲喂与管理
试验在宁夏盐池滩羊育种选育场进行。分别选择6月龄健康和具有良好系谱的滩羊公羊163只,母羊182只。试验采用单栏颈夹饲喂,在采食期间采用颈夹固定羔羊,采食完成后松开颈夹,让其自由运动[11]。试验饲粮配方和加工饲粮营养水平参考肉羊饲养标准[12],试验饲粮按配方加工为颗粒饲料。日粮组成及营养水平与陈丽尧等[13]所报道一致(表1)。试验过渡期15 d,正试期50 d,试验期间饲喂同一种饲料。在试验开始前记录绵羊初始体重,并且每10 d称重一次至试验结束。
表 1 基础日粮组成及营养水平Table 1. Composition and nutrient levels of basal diets项目 Item 含量 Content 原料 Ingredient/% 玉米秸秆 Corn straw 20.0 玉米 Corn 32.0 糖蜜 Molasses 4.0 豆粕 Soybean meal 6.0 棉粕 Cotton meal 8.0 玉米皮 Corn bran 15.3 玉米胚芽粕 Corn germ meal 11.0 石粉 Limestone 1.2 食盐 NaCl 0.5 预混料 Premix 2.0 合计 Total 100.0 营养水平(干物质基础)
Nutrient level (Dry matter basis)1)代谢能 Metabolic energy/(MJ·kg−1) 9.93 粗蛋白质 Crude protein/% 13.74 粗脂肪 Ether extract/% 2.74 中性洗涤纤维 Neutral detergent fibers/% 35.99 酸性洗涤纤维 Acid detergent fibers/% 20.34 钙 Calcium/% 0.71 磷 Phosphorus/% 0.31 1)预混料为每千克饲粮提供:铁69.63 mg,铜7.41 mg,锰23.7 mg,锌55 mg,碘0.67 mg,钴0.3 mg,维生素A 2500 IU,维生素E 23 IU。2)代谢能为计算值,其他营养水平为测定值。
1)Premix provided the following per kilogram of the diet: Fe 69.63 mg, Cu 7.41 mg, Mn 23.7 mg, Zn 55 mg, I 0.67 mg, Co 0.3 mg, VA 2 500 IU, and VE 23 IU. 2)Metabolic energy calculated value, nutrient levels were measured values.1.2 RFI表型计算
RFI 通过回归模型计算,所用模型[13]如下:
$ Y_{i }= \beta_{0} + \beta_{1} Mid{\text{-}} BW_{i} ^{0.75} + \beta_{2} ADG_{i} + e_{i}。 $
式中:Yi为第i只动物的DMI(干物质采食量);β0为回归截距;β1为Mid-BW 0.75对应的回归系数;β2为ADG (日增重)对应的回归系数;ei为第i只动物的随机误差,Mid-BWi0.75
为第i只动物的中期代谢体重,ADGi为第i只动物的平均日增重。 公、母滩羊的回归方程依次为:
DMI预测 = − 0.244 + 1.669Mid-BW0.75 + 0.079ADG。
DMI预测 = 0.103 + 2.126 × Mid-BW0.75 + 0.051ADG。
RFI = RFI实际 – RFI预测。
当个体RFI ≥ RFI平均值+0.5时,划分为高RFI组;当个体RFI ≤ RFI平均值–0.5时,划分为低RFI组。
1.3 瘤胃液采集
饲养试验结束后选取极高RFI母羊和公羊各7只,极低RFI母羊和公羊各8只。在第66天晨饲前采用绵羊瘤胃导管体外抽取瘤胃液,并将瘤胃液分管装入50 mL冻存管,置于−20 ℃条件下保存,用于瘤胃微生物DNA的提取。
1.4 瘤胃液DNA 提取
采用CTAB法[9]提取样本基因组DNA,然后利用琼脂糖凝胶电泳和NanoDrop 2000分光光度计检测DNA纯度和浓度,取适量样品于离心管中,使用无菌水稀释样品至1 ng·µL−1。
1.5 16S rDNA V4区PCR扩增子测序
根据测序区域选择,使用带Barcode的特异引物。16S V4区域引物为515F-806R,上游引物F:CCTAYGGGRBGCASCAG/下游引物R:GGACTACNNGGGTATCTAAT。使用New England Biolabs公司的Phusion® High-Fidelity PCR Master Mix with GC Buffer。对高效和高保真的酶进行PCR,确保扩增效率和准确性。PCR产物使用2%浓度的琼脂糖凝胶进行电泳检测。根据PCR 产物浓度进行等浓度混样,充分混匀后使用1 × TAE 浓度2%的琼脂糖胶电泳纯化PCR 产物,选择主带400~450 bp的序列,割胶回收目标条带。使用Illumina公司TruSeq DNA PCR-Free Library Preparation Kit建库试剂盒进行文库的构建,构建好的文库经过Qubit定量和文库检测,合格后,使用NovaSeq 6000进行上机测序。
1.6 信息分析与数据统计
剔除DNA降解或不合格样本后,获得极高组RFI母羊6只和公羊5只、极低组RFI母羊6只和公羊7只的原始数据。测序得到的原始数据,存在一定比例的干扰数据,为了使信息分析的结果更加准确可靠,对原始数据进行拼接过滤,得到有效数据。基于有效数据进行OTUs (operational taxonomic units)聚类和物种分类分析,根据OTUs聚类结果,对每个OTUs的代表序列做物种注释,得到对应的物种信息和基于物种的丰度分布情况。同时,对OTUs进行丰度、Alpha多样性计算、Venn图,以得到样品内物种丰富度和均匀度信息、不同样品或分组间的共有和特有OTUs信息。对OTUs进行多序列比对并构建系统发生树,进一步得到不同样品和分组的群落结构差异,通过主坐标分析(PCoA)和样品聚类树进行展示。为进一步挖掘分组样品间的落结构差异,选用T-test和LEfSe统计分析方法对分组样品的物种组成和群落结果进行差异显著性检。
利用Uparse软件(Uparse v7.0.1001,http://www.drive5.com/uparse/)对所有样本的全部 Effective Tags进行聚类,默认以97%的一致性将序列聚类成为OTUs。对OTUs序列进行物种注释,用Mothur方法与SILVA138 (http://www.arb-silva.de/)的SSUrRNA数据库进行物种注释分析(设定阈值为0.8~1)。使用MUSCLE (Version 3.8.31,http://www.drive5.com/muscle/)软件进行快速多序列比对,得到所有OTUs代表序列的系统发生关系。使用Qiime软件(Version 1.9.1)计算Observed-OTUs,Chao1,Shannon,Simpson,ace,Goods-coverage,PD_whole_tree指数,使用R软件(Version 2.15.3)绘制稀释曲线、rank abundance曲线、物种累积曲线,并使用R软件进行α多样性指数组间差异分析;α多样性指数组间差异分析进行非参数检验,选用agricolae包的wilcox检验。试验数据使用 Excel 2010 进行整理,应用SPSS 17.0 进行独立样本 t 检验,P < 0.05时认为差异显著,结果均以平均值 ± 标准误(means ± SEM)形式列出。PCoA基于unweighted unifrac 距离来分析。
2. 试验结果
2.1 菌群测序量及序列长度分布
对瘤胃液样本的肠道菌群DNA片段进行双端测序,筛选过滤后共产生2 063 628条原始读段,平均每个样本85 970条原始读段。经质控筛选过滤后共产生2 028 835条有效读段,平均每个样本84 535条有效读段。其中Q20和Q30 (碱基质量值≥20或30所占的比例)平均值分别为98%和94% (附表1)。
2.2 OUT聚类分析
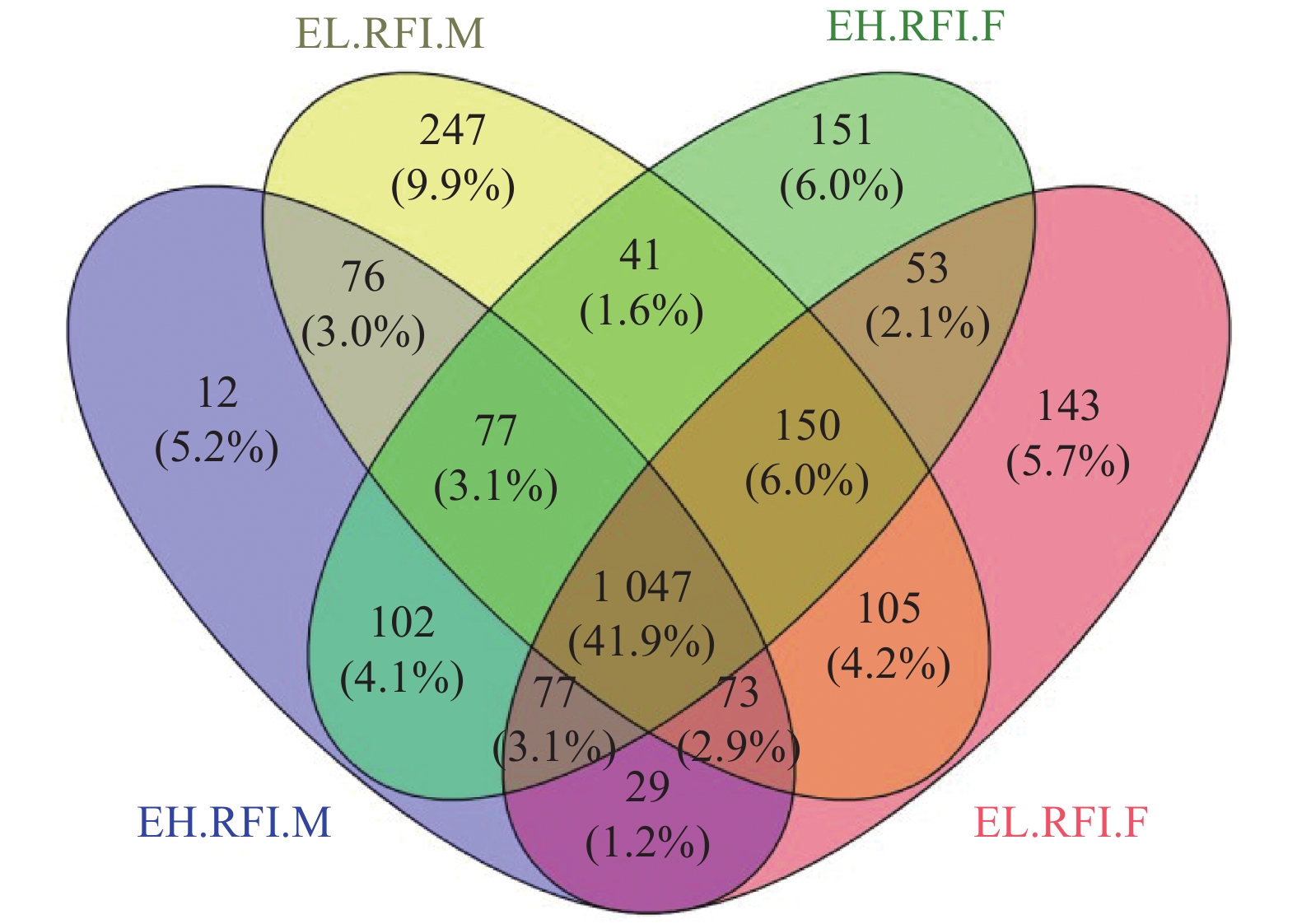
OTUs是人为设置的微生物分类单元,按照序列相似度≥97%归为同一个OTU,即同一微生物物种的标准进行聚类,经测序后,基于97%物种相似性,将24组瘤胃液样品中获得的序列进行OTU聚类,对样品进行OTU的统计,剔除稀有OTU后,Venn图显示(图1),极低组公羊发现1 812个OTUs,极高组公羊发现1 610个OTUs,极低组母羊发现1 677个OTUs,极高组母羊发现1 698个OTUs。极高组公羊和极低组公羊两组共有1 274个OTUs。极高组母羊和极低组母羊共有1 327个OTUs,极高组公羊和母羊共有1 303个OTUs,极低组公羊和母羊共有1 377个OTUs。
![]() 图 1 不同RFI组OTUsVenn图EHF、EHM、ELF和ELM分别表示极高组母羊、极高组公羊、极低组母羊、极低组公羊;下同。Figure 1. OTUs Venn diagram of different RFI groupsEHF, EHM, ELF, and ELM indicate extremely high groups of females and males, and low groups of females and males, respectively. This is applicable for the following tables and figures as well.
图 1 不同RFI组OTUsVenn图EHF、EHM、ELF和ELM分别表示极高组母羊、极高组公羊、极低组母羊、极低组公羊;下同。Figure 1. OTUs Venn diagram of different RFI groupsEHF, EHM, ELF, and ELM indicate extremely high groups of females and males, and low groups of females and males, respectively. This is applicable for the following tables and figures as well.2.3 微生物群落Alpha多样性分析
所有组观测到的物种数、Shannon-Wiener指数、Simpson指数、Chao1指数、ACE指数和基因文库覆盖率均无显著差异(P > 0.1),EHM与ELM 的PD_whole_tree差异显著(P < 0.1)(表2)。但是极高组公羊的PD_whole_tree 明显高于其他组别。表明极高低组RFI公羊和极高低组母羊之间菌群多样性差异较小,菌群趋于一致性。
表 2 不同剩余采食量对滩羊瘤胃微生物多样性影响Table 2. Different residual feed intake on rumen microbial diversity of Tan sheep项目
Item组别 Group P EHM ELM EHF ELF 观测到的物种数 No. of observed species 860.00 ± 27.37 838.00 ± 41.78 857.00 ± 68.35 837.00 ± 55.81 0.983 Shannon-Wiener 5.79 ± 0.35 6.33 ± 0.14 6.02 ± 0.49 6.01 ± 0.32 0.724 Simpson 0.91 ± 0.22 0.96 ± 0.01 0.94 ± 0.21 0.94 ± 0.14 0.265 Chao1 961.04 ± 30.71 926.89 ± 39.40 961.99 ± 65.24 937.14 ± 58.35 0.947 ACE 975.04 ± 29.93 947.22 ± 39.42 984.36 ± 67.55 962.56 ± 56.61 0.956 覆盖率 Good’s_coverage 0.996 0 ± 0.000 4 0.996 0 ± 0.000 1 0.996 0 ± 0.000 3 0.996 0 ± 0.000 2 0.573 PD_whole_tree 71.87 ± 2.33b 102.23 ± 11.78a 74.92 ± 7.73b 74.44 ± 6.86 0.058b 同行不同大小写字母分别表示在0.05和0.1水平上差异显著。下表同。
Different capital and lowercase letters indicate significant differences at the 0.05 and 0.1 levels, respectively. this is appicable for the following tables as well.2.4 微生物群落多样性曲线
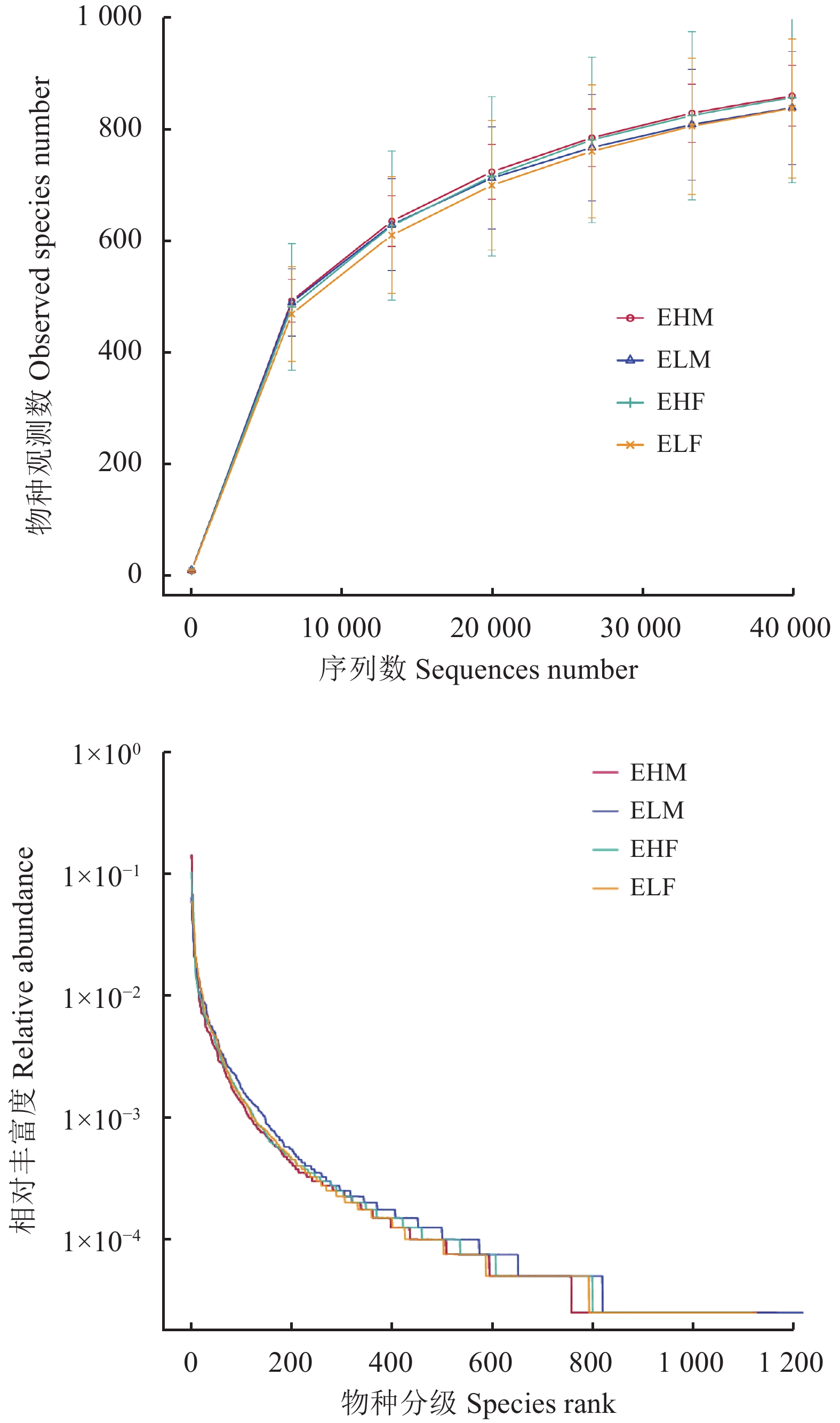
当测序量达到3 500时,不同RFI组物种曲线趋向平坦,说明测序数据量渐合理。等级聚类曲线可直观地反映样本中物种的丰富度和均匀度。在水平方向上,物种的丰富度逐渐变宽,物种的丰富度升高,曲线在横轴上的跨度越大;在垂直方向上,曲线逐渐变平缓,物种分布均匀性良好(图2)。
2.5 微生物群落β多样性分析
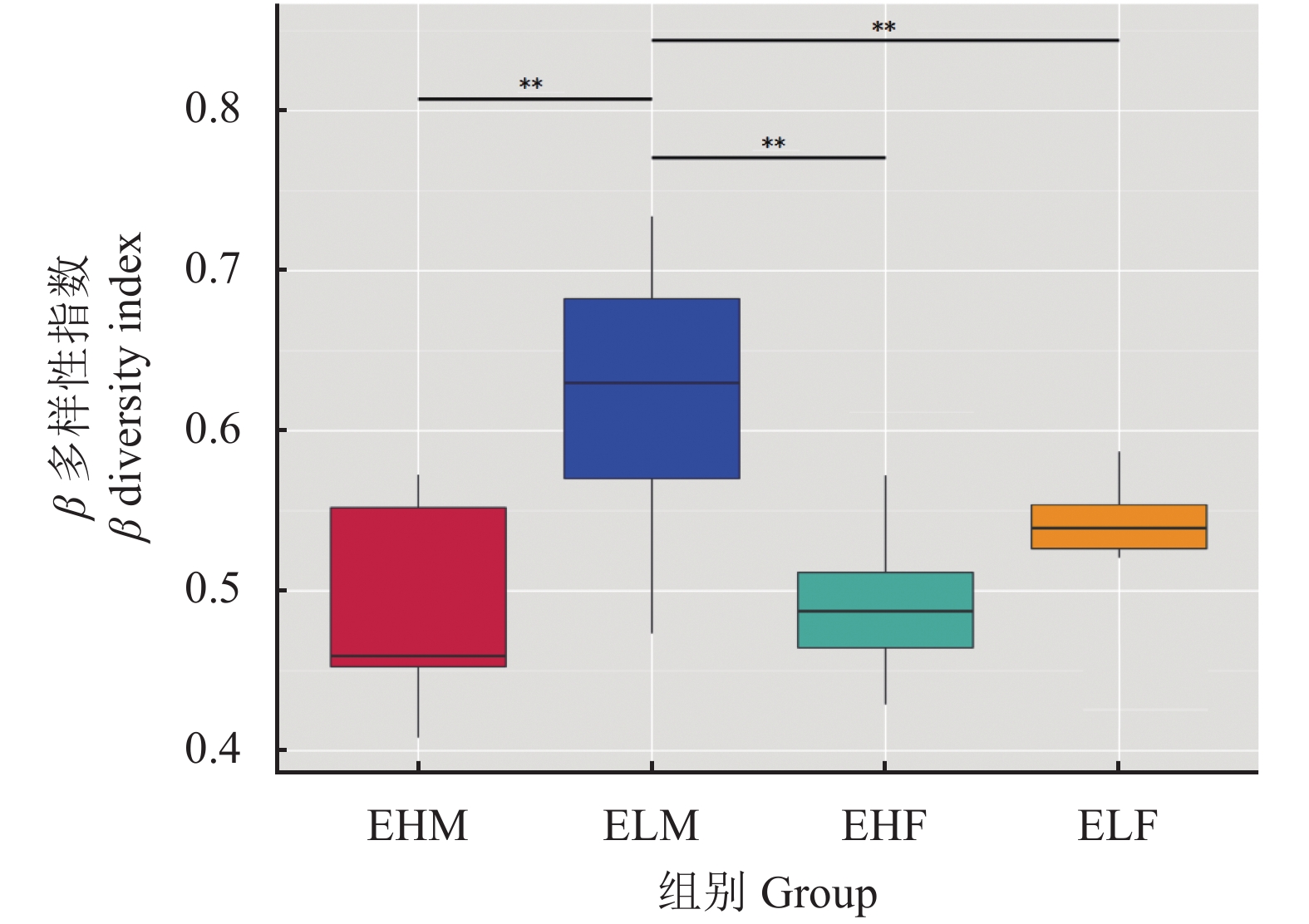
β多样性组间差异分析的箱型图(图3)表明,公羊极高组与极低组之间存在极显著差异(P < 0.01),母羊极高组与极低组之间差异不显著(P > 0.05)。极低组公羊和母羊之间差异极显著(P < 0.01),极高组公羊和母羊之间差异不显著(P > 0.05)。
2.6 主坐标分析
滩羊不同RFI组间样本具有部分重叠,但没有明显的分离,表明样本组间和组内微生物群落差异性较小(图4)。
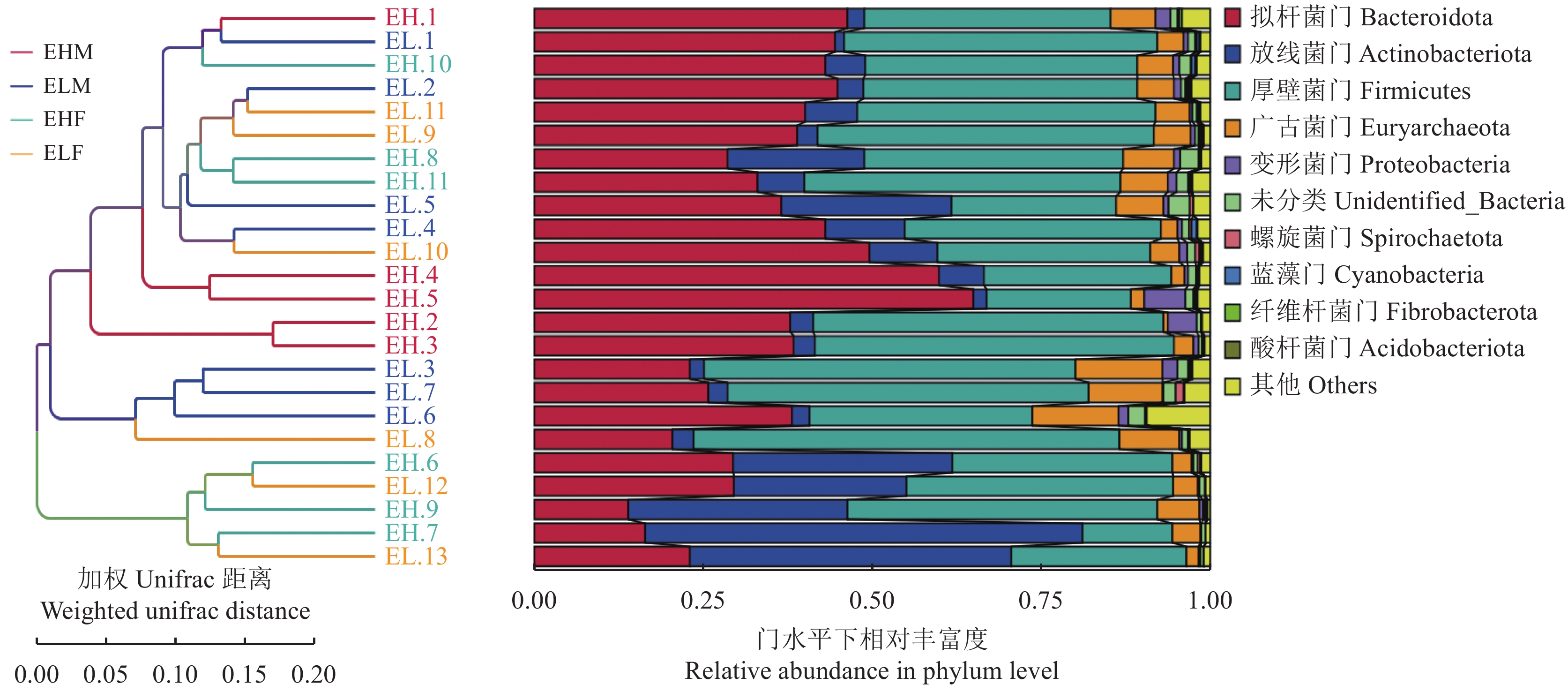
2.7 UPGMA聚类
极高RFI组公羊4个样本聚集在一起,聚类效果较好,其他RFI组样本聚类效果不明显(图4)。从整体结构来看,不同RFI组微生物结构相似。
2.8 滩羊瘤胃微生物组成
滩羊瘤胃微生物在门和属水平上公母羊优势菌属不同,但是整体结构相似。在门水平上,公羊优势菌门是拟杆菌门和厚壁菌门,母羊优势菌门是厚壁菌门和拟杆菌门 (表3)。在属水平上,公羊优势菌属为普雷沃氏菌属(Prevotella)、韦荣球菌UCG-002 (Erysipelotrichaceae_UCG_002),母羊优势菌属是Olsenella和普雷沃氏菌属(表4)。
在门水平上(表3),拟杆菌门公羊极高组与极低组之间差异显著(P < 0.05),极高组公羊显著高于母羊。放线菌门公羊极低组明显高于极高组,极高组母羊明显高于公羊。广古菌门公羊极高组显著低于极低组(P < 0.05)。变形菌门极高组公羊显著高于其他组(P < 0.05)。在属水平上(表4),Olsenella 菌属极高组母羊明显高于公羊,极低组公羊明显高于母羊。韦荣球菌科UCG-002极高组公羊显著高于其他组别(P < 0.05)。甲烷短杆菌极高组公羊与极低组公羊明显高于其他组别。
表 3 微生物相对丰度在门分类水平上排名前十的物种Table 3. Top 10 species at phylum taxonomic level in relative abundance of microorganisms门
Phlyum组别 Group P EHM ELM EHF ELF 拟杆菌门 Bacteroidota 49.58 ± 5.56A 36.66 ± 3.38B 27.48 ± 4.42B 33.67 ± 4.57B 0.022 放线菌门 Actinobacteriota 3.51 ± 0.81c 7.05 ± 3.30b 27.10 ± 8.90a 16.15 ± 7.12ab 0.057 厚壁菌门 Firmicutes 38.08 ± 6.35 41.47 ± 4.16 36.11 ± 5.05 42.27 ± 5.41 0.811 广古菌门 Euryarchaeota 2.81 ± 1.01B 7.95 ± 1.62A 5.55 ± 0.73A 4.88 ± 0.95A 0.050 变形菌门 Proteobacteria 2.80 ± 1.07A 0.99 ± 0.26B 0.70 ± 0.18B 0.54 ± 0.13B 0.019 未分类 Unidentified_Bacteria 0.88 ± 0.13 1.65 ± 0.33 1.27 ± 0.37 0.73 ± 0.12 0.115 螺旋菌门 Spirochaetota 0.15 ± 0.05 0.43 ± 0.14 0.14 ± 0.05 0.17 ± 0.08 0.115 蓝藻门 Cyanobacteria 0.15 ± 0.06 0.24 ± 0.10 0.18 ± 0.08 0.19 ± 0.06 0.592 纤维杆菌门 Fibrobacterota 0.11 ± 0.07 0.11 ± 0.03 0.08 ± 0.05 0.04 ± 0.02 0.631 酸杆菌门 Acidobacteriota 0.06 ± 0.02 0.05 ± 0.02 0.09 ± 0.03 0.08 ± 0.04 0.426 表 4 微生物相对丰度在属分类水平上排名前十的物种Table 4. Top ten species at genus taxonomic level in relative abundance of microorganisms属
Genus组别 Group P EHM ELM EHF ELF Olsenella 2.89 ± 0.80b 4.66 ± 2.06b 23.66 ± 8.84a 13.84 ± 6.80a 0.069 普雷沃氏菌属 Prevotella 32.66 ± 5.05a 20.44 ± 4.09ab 16.00 ± 4.00b 16.83 ± 9.23b 0.057 韦荣球菌属 UCG_002 Erysipelotrichaceae_UCG_002 14.22 ± 7.41A 0.26 ± 0.05B 3.37 ± 2.40B 0.10 ± 0.02B 0.028 Quinella 0.02 ± 0.00 1.73 ± 1.65 0.42 ± 0.10 5.10 ± 4.76 0.522 月形单胞菌属 Selenomonas 3.91 ± 1.79ab 8.21 ± 2.80a 1.73 ± 0.43b 2.68 ± 1.27b 0.094 甲烷短杆菌 Methanobrevibacter 2.77 ± 0.10b 7.82 ± 1.62a 5.51 ± 0.72b 4.81 ± 0.95b 0.055 双歧杆菌属 Bifidobacterium 0.34 ± 0.05 2.06 ± 1.50 2.84 ± 1.31 1.73 ± 0.52 0.531 Acetitomaculum 1.58 ± 0.25 2.96 ± 1.38 2.93 ± 1.09 3.79 ± 1.12 0.634 瘤胃球菌属 Ruminococcus 2.13 ± 0.77 4.12 ± 1.29 1.69 ± 0.43 2.14 ± 0.82 0.216 毛螺菌 NK3A20 Lachnospiraceae_NK3A20_group 1.85 ± 1.24 3.90 ± 1.01 3.28 ± 0.68 3.36 ± 0.66 0.420 2.9 差异微生物的LEfSe及功能富集分析
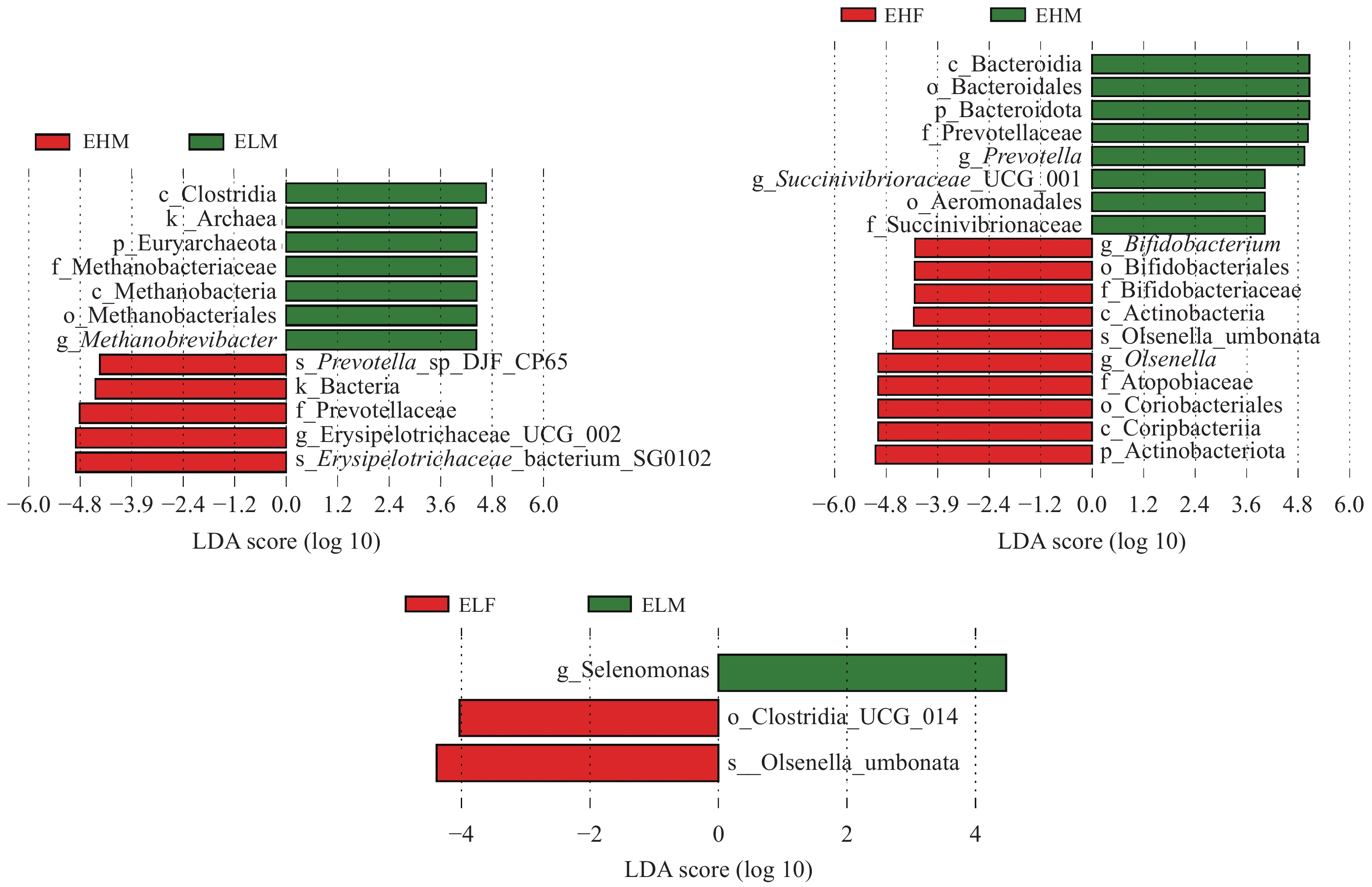
LEfSe可以在各个水平上比较微生物丰度差异,以线性判别分析(LDA) 得分 > 4为符合生物标记物的菌落。极高组公羊与极低组公羊比较发现梭菌纲、古菌域、广古菌门、甲烷杆菌科、甲烷杆菌纲、甲烷杆菌目和甲烷短杆菌属(Methanobrevibacter)在极低组公羊中高表达,Prevotella_DJF_cp65、Bacteria、普雷沃氏菌科、韦荣球菌科UCG-002 和韦荣球菌科SG0102在极高组公羊中高表达(图5)。极高组母羊和极高组公羊比较发现, Bacteroidia、Bacteroidales、Bacteroidota、Prevotellaceae、Prevotella、Succinivibrionaceae_UGG_001、Aeromonadales、Succinivibrionaceae在极高组公羊中高表达,双歧杆菌属(Bifidobacterium)、双歧杆菌目、双歧杆菌科、放线菌纲、Olsenella. umbonata、Olsenella、Atopobiaceae、Coriobacteriales、Coriobacteriia和Actinobacteriota在极高组母羊中高表达(图5)。比较发现,月形单胞菌属 (Selenomonas)在极低组公羊中高表达,梭状芽胞杆菌UGG.014 (Clostridia_UGG_014)、Olsenella_umbonata在极低组母羊中高表达(图6)。
3. 讨论
本研究选择较大的滩羊群体,具有较高的生物学重复性,并且选择公母羊两类群体作为研究对象,探究性别差异差异对瘤胃微生物的影响。Zhang等[14]在湖羊饲料效率的研究中采用单栏饲喂的方式,而这无疑忽略了动物运动对机体造成的影响。研究发现,肉牛身体活动量与RFI表型相关系数高达0.32,其中10%的RFI变异由身体活动所导致,这些活动包括采食、反刍以及物理性运动[15]。在动物不同生长阶段,性别对生物发育速度具有较大的影响,动物生长初期雌性动物的生长发育速度较快,而在生物生长发育后期,雄性动物的生长发育速度则较快[16-17],因此动物性别也可能会对饲料效率有影响。
3.1 性别差异对微生物菌群的影响
近年来发现性别是影响机体代谢的重要因素。Verschuren等[18]报道了性别是影响不同饲料效率猪肠道微生物的重要影响因素。Ahmed等[3]报道了安格斯牛性别差异对饲料效率的影响,研究发现性别对不同RFI动物生长性能和瘤胃微生物无显著影响,但对瘤胃上皮基因表达差异显著。在本研究中,极低组母羊和公羊PD_whole_tree差异明显,而其他公母滩羊性别对不同RFI瘤胃微生物菌群α多样性无显著影响。通过不同性别RFI滩羊PCoA和NMDS图分析发现,各组别差异较小,β多样性差异不显著。α和β多样性指数是反映样本微生物丰富度和均匀度的两个重要指标。这表明性别对微生物生长影响较小。从微生物组成来看,在门水平上,不同性别组别间优势菌群种类一致,极高组公母羊拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)和变形菌门(Proteobacteria)差异显著。在属水平上,公母羊间第1优势菌属发生变化,Olsenella菌属为母羊第1菌属,普雷沃氏菌属(Prevotella)为公羊第1优势菌属。极高组公母羊Olsenella、普雷沃氏菌属和韦荣球菌属UGG-002 (Erysipelotrichaceae_UCG_002)差异显著。极低组公母羊月单胞菌属(selenomonas)差异显著。韩学平等[19]研究了不同性别间牦牛瘤胃微生物多样性,发现公母牦牛间微生物组成在种属间组成一致,而在本研究中发现,不同RFI母滩羊在属水平下优势种属发生了变化。因此Olsenella菌属可能与母羊RFI有关,不同RFI公母羊具有不同的优势,可能影响瘤胃微生物的不同发酵方式。
3.2 不同RFI微生物菌群差异
瘤胃是反刍动物的主要消化器官,栖居大量的微生物,并具有复杂的微生物生态系统。研究发现,微生物菌群的变化直接影响动物瘤胃发酵和营养物质的吸收,进而导致不同饲料效率的差异[20-21]。为此,对不同RFI滩羊瘤胃微生物进行了研究。本研究选择16S V3-V4,341F-806R区域,分析瘤胃内微生物多样性。
本研究中,序列有效读取率为98%,数据具有较高的完整性。其中极低RFI公羊具有较高的OTUs数目,公羊极高组(1 812)极低组(1 610) OTUs数目相差较大,极低组高于极高组,而母羊极高极低组OTUs数目差异不显著,极高组高于极低组。马万浩等[22]报道发现,不同RFI湖羊公羊中OTUs数目高RFI组(394)高于低RFI组(353)。造成这种差异的因素有很多,例如精粗比,随着精料上升,菌群数量下降,也可能是动物品种和饲喂环境的原因。α和 β多样性指数是研究微生物多样性的两个重要参数。本研究发现,α多样性中物种数、Chao1、ACE 值、Shannon指数、Simpson指数和覆盖率等参数在4个群体间没有差异,其中只有PD_whole_tree在极高和极低RFI公羊中差异显著,通过柱状图、PCoA图和UPGMA等没有聚类和分离。这与马万浩等[22]研究结果一致,同样与Myer等[23]通过肉牛平均日采食量和动物体重来评估饲料效率研究结果一致,所有样本和类群发现观察到的OTUs、丰富度(Chao1)或多样性没有差异。这表明每个类群内微生物群落之间的相似性。
在门分类学水平上,4个组别具有相同的优势菌门,分别是拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes),拟杆菌门差异显著,这与众多研究结果一致[24-26],其优势菌门差异的变化主要发生在厚壁菌门(Ruminococcus、Lachnospira、Lachnospira_NK3A20、Erysipelotrichaceae_UCG-002、Quinella、Selenomonas菌属中),Myer等[23]恰好证明厚壁菌门的增加与ADFI较高的动物有关,这可能与饲料效率有关。Erysipelotrichaceae_UCG-002和Selenomonas主要参与发酵代谢类型,参与丙酮酸、乳酸发酵和葡萄糖发酵主要产生乙酸和丙酸,为机体供能。在本研究结果中极高和极低母羊组中具有较高的放线菌门中的菌属(Olsenella、Bifidobacterium),Bifidobacterium可以对葡萄糖进行异型乳酸发酵,生成L ( + )乳酸和乙酸(摩尔比为3 ꞉ 2),不产CO2,经常作为益生菌的调节剂[27]。Perea等[28]的研究表明,不同RFI的阉羊在瘤胃内纤维杆菌、瘤胃球菌、糖酵母菌、双歧杆菌、变形杆菌存在显著差异。占今舜等[29]报道,随着精料比例增加瘤胃中放线菌门数量增加。研究报道,拟杆菌门丰度的高低可能与高纤维饲粮有关,拟杆菌门又是降解碳水化合物的组要微生物群[30]。本研究中,极高组公羊与极高组母羊拟杆菌门差异显著,高RFI公羊具有较高的拟杆菌门,因此高RFI公羊具有较高消化纤维类饲料能力。在公羊不同RFI表型中广古菌门(Euryarchaeota)中的甲烷短杆菌(Methanobrevibacter)和变形菌门差异显著。广古菌门在动物胃肠道中主要以产甲烷菌的形式存在,Methanobrevibacter通常将无机或有机化合物厌氧发酵转化成甲烷和二氧化碳排放,造成巨大的能量损失和环境污染。众多研究发现,甲烷排放与饲料效率有关[31-33],Ellison等[34]发现采用青贮料饲喂羔羊时,Methanobrevibactr在低RFI羊瘤胃中丰度更高,这与本研究结果一致。然而,也有研究报道,瘤胃内产甲烷菌的丰度在高RFI动物中更高,这似乎更符合理论事实[35]。造成这种原因的结果可能与其他菌群相互作用有关,董利锋等[36]报道,Geobacterspp的存在能够直接通过电子转移与产甲烷菌形成协同作用,减少有机物发酵产物的积累而促进产甲烷菌的定植。Geobacterspp是属于变形菌门Geobacteraceae科的微生物,恰好在本研究结果中发现,两种变形菌门菌属Olsenella、Bifidobacterium,并且Olsenella和Bifidobacterium在极高RFI母羊中的丰度均高于极低RFI母羊,因此Olsenella和Bifidobacterium具有与Geobacterspp类似的作用。
4. 结论
性别对极高或极低RFI个体瘤胃微生物有显著影响,Olsenella菌属为母羊第1菌属,普雷沃氏菌属(Prevotella)为公羊第1优势菌属异,但不同RFI 滩羊群体间瘤胃菌群多样化和丰富度相似,瘤胃微生态环境相对稳定。优势菌群的种类和丰度在不同 RFI 组滩羊瘤胃微生物区系中基本一致,厚壁菌门菌群数量的增加可能与RFI有关;不同RFI公羊组广古菌门中的Methanobrevibacter菌群在低RFI羊瘤胃中丰富度更高,不同RFI母羊组微生物多样性差异不显著。
参考文献
[1] KOCH R M, SWIGER L A, CHAMBERS D, GREGORY K E. Efficiency of feed use in beef cattle. Journal of Animal Science, 1963, 22(2): 486-494. doi: 10.2527/jas1963.222486x
[2] HERD R M, ARTHUR P F. Physiological basis for residual feed intake. Journal of Animal Science, 2009, 87 (14 Suppl): 64-71.
[3] AHMED A, ELOLIMY, MOHAMED K, ABDELMEGEI D, JOSHUA C, MCCANN, DANIL W, SHIKE, JUAN J L. Residual feed intake in beef cattle and its association with carcass traits, ruminal solid-fraction bacteria, and epithelium gene expression. Journal of Animal Science and Biotechnology, 2018(9): 67.
[4] PACHECO D, WAGHORN G, JANSSEN P H. Decreasing methane emissions from ruminants grazing forages: A fit with productive and financial realities. Animal Production Science, 2014, 54(9): 1141-1154. doi: 10.1071/AN14437
[5] GUAN L L, NRUMAH J D, BASARAB J A, MOORE S S. Linkage of microbial ecology to phenotype: Correlation of rumen microbial ecology to cattle’s feed efficiency. FEMS Microbiology Letters, 2008, 288(1): 85-91. doi: 10.1111/j.1574-6968.2008.01343.x
[6] DÉBORAH R G L, MÁRCIO D S D, ALEX J L R. Assessing the relationship between the rumen microbiota and feed efficiency in Nellore steers. Journal of Animal Science and Biotechnology, 2021, 12(4): 1645-1661.
[7] ZHANG Y K, ZHANG X X, LI F D, LI C, ZHANG D Y, SONG Q Z, LI X L, ZHAO Y, WANG W M. Characterization of the rumen microbiota and its relationship with residual feed intake in sheep. Animal, 2021, 15(3): 100161. doi: 10.1016/j.animal.2020.100161
[8] ELLISON M J, CONANT G C, LAMBERSON W R, COCKRUM R R, AUSTIN K J, RULE D C, CAMMACK K M. Diet and feed efficiency status affect rumen microbial profiles of sheep. Small Ruminant Research, 2017, 156(11): 12-19.
[9] CLEMONS B A, SHIN S B, SMITH T P L, EMBREE M M, VOY B H, SCHNEIDER L G, DONOHOE D R, MCLEAN Kyle J, MYER P R. Ruminal protozoal populations of angus steers siffering in feed efficiency. Animals, 2021, 11(6): 1561. doi: 10.3390/ani11061561
[10] LIANG Y, LI G, LI X, LU J, LI F, TANG D. Growth performance, rumen fermentation, bacteria composition, and gene expressions involved in intracellular ph regulation of rumen epithelium in finishing Hu lambs differing in residual feed intake phenotype. Journal of Animal Science, 2017, 95(4): 1727-1738.
[11] 陶金忠, 和东迁, 王俊奎, 陈丽尧, 罗芳, 张倩, 卢童童, 吴少飞. 一种滩羊单栏饲喂设备: ZL 213603732U, 2021-07-06. TAO J Z, HE D Q, WANG J K, CHEN L Y, LUO F, ZHANG Q, LU T T, WU S F. The utility model relates to a single stall feeding device for Tan sheep: ZL 213603732U, 2021-07-06.
[12] 中华人民共和国农业部. NY/T816–2004 肉羊饲养标准. 北京: 中国标准出版社, 2004. Ministry of Agriculture of the People's Republic of China. NY/T816–2004 Feeding Standad of Meat-Producing Sheep and Goats. Beijing: China Standard Press, 2004.
[13] 陈丽尧, 和东迁, 卢童童, 张倩, 吴少飞, 罗芳, 王俊奎, 张瑞雪, 陶金忠. 在不限定运动的情况下滩羊RFI的测定及与其他生长性能之间的关系. 中国兽医学报, 2022, 42(1): 2056-2061. CHEN L Y, HE D Q, LU T T, ZHANG Q, WU S F, LUO F, WANG J K, ZHANG R , TAO J Z. Masurement of RFI traits of Tan sheep and the relationship with other growth perfprmance without restricting exercise. Chinese Journal Veterinay Science, 2022, 42(1): 2056-2061.
[14] ZHANG X X, LI G Z, LI F D, ZHANG D Y, YUAN L F, ZHAO Y, ZHANG Y K, LI X L, SONG Q Z, WANG W M. Effect of feed efficiency on growth performance, body composition, and fat deposition in growing Hu lambs. Animal Biotechnology, 2021(11): 1-16.
[15] CHEN L Y, HE D Q, LU T T, ZHANG Q, WU S F, LUO F, WANG J K, ZHANG R X, TAO J Z. Measurement of RFI of Tan sheep without restricted exercise and its relationship with other growth performance. Chinese, 2022, 42(1): 6.
[16] LI F, LE L G. Metatranscriptomic profiling reveals linkages between the active rumen microbiome and feed efficiency in beef cattle. Applied and Environmental Microbiology, 2017, 83(9): e00061-17.
[17] 石婧, 姚勤, 陈克平. 性别对生物生长发育速率影响的研究. 安徽农业科学, 2011, 39(22): 13301-13302, 13305. doi: 10.3969/j.issn.0517-6611.2011.22.018 SHI J, YAO Q, CHEN K P. Effects of sex on biological growth and development rate. Journal of Anhui Agricultural Sciences, 2011, 39(22): 13301-13302, 13305. doi: 10.3969/j.issn.0517-6611.2011.22.018
[18] VERSCHUREN L M G, CALUS M P L, JANSMAN A J M, BERGSMA R, KNOL E F, GILBERT H, ZEMB O. Fecal microbial composition associated with variation in feed efficiency in pigs depends on diet and sex. Journal of Animal Science, 2018, 96(9): 1405-1418.
[19] 韩学平, 刘宏金, 胡林勇, 徐世晓, 艾德强, 付弘贇, 陈永伟, 赵建忠. 环湖牦牛瘤胃微生物区系特征及性别之间的差异. 动物营养学报, 2020, 32(1): 234-243. doi: 10.3969/j.issn.1006-267x.2020.01.029 HAN X P, LIU H J, HU L Y, XU S X, AI D Q, FU H Y, CHEN Y W, ZHAO J Z. Characteristics and gender differences of rumen microflora in yaks around lake. Chinese Journal of Animal Nutrition, 2020, 32(1): 234-243. doi: 10.3969/j.issn.1006-267x.2020.01.029
[20] ELIE J, WHITE B A, ITZHAK M, HEIMESAAT M M. Potential role of the bovine rumen microbiome in modulating milk composition and feed efficiency. PLoS One, 2014, 9(1): e85423. doi: 10.1371/journal.pone.0085423
[21] PATIL R D, ELLISON M J, WOLFF S M, SHEARER, COURTNEY, WRIGHT A, COCKRUM R, AUSTIN K, LAMBERSON W, CAMMACK K, CONANT G. Poor feed efficiency in sheep is associated with several structural abnormalities in the community metabolic network of their ruminal microbes. Journal of Animal Science, 2018, 96(6): 2113-2124. doi: 10.1093/jas/sky096
[22] 马万浩, 梁玉生, 张智安, 董春晓, 李飞, 李发弟, 翁秀秀. 不同剩余采食量的湖羊生长性能和消化道微生物多样性差异的研究. 动物营养学报, 2019, 31(9): 4080-4091. MA W H, LIANG Y S, ZHANG Z A, DONG C X, LI F, LI F D, WENG X X. Difference of grobial diversity of Hu sheep with different residual feed intake. Chinese Journal of Animal Ntrition, 2019, 31(9): 4080-4091.
[23] MYER P R, SMITH T, WELLS J E, KUEHN L A, FREETLY H C, FORSTER R J. Rumen microbiome from steers differing in feed efficiency. PLoS One, 2015, 10(6): e0129174. doi: 10.1371/journal.pone.0129174
[24] WANG W, LI C, LI F, WANG X, ZHANG X, LIU T. Effects of early feeding on the host rumen transcriptome and bacterial diversity in lambs. Scientific Reports, 2016, 6(8): 32479.
[25] LI F, LI C, CHEN Y, MALLORY M E, BRYNN H V, LIESEL G S, DALLAS R D, KYLE J M, PHILIIP R M. Host genetics influence the rumen microbiota and heritable rumen microbial features associate with feed efficiency in cattle. Microbiome, 2019, 7(1): 92. doi: 10.1186/s40168-019-0699-1
[26] 张小雪. 不同剩余采食量羔羊生产性能和瘤胃微生物区系及肝脏转录组研究. 兰州: 兰州大学博士学位论文, 2019. ZHANG X X. Study on performance, rumen microflora and liver transcriptome of lambs with different residual feed intake. PhD Thesis. Lanzhou: Lanzhou University, 2019.
[27] 孙纪录, 贾英民, 高小芹, 田洪涛, 张伟. 菊芋在体内对双歧杆菌生长的影响研究. 食品科技, 2007, 32(1): 212-214. doi: 10.3969/j.issn.1005-9989.2007.01.064 SUN J L, JIA Y M, GAO X Q, TIAN H S, ZHANG W. Effects of Jerusalem artichos on the growth of bifidobacteria in vivo. Food Science and Technology, 2007, 32(1): 212-214. doi: 10.3969/j.issn.1005-9989.2007.01.064
[28] PEREA K, PERZ K, OLIVO S K, WILLIAMS A, YEOMAN C J. Feed efficiency phenotypes involve changes in ruminal, colonic, and small intestine-located microbiota. Journal of Animal Science, 2017, 95(6): 2585-2592.
[29] 占今舜, 杨群, 胡耀, 武艳平, 霍俊宏. 日粮精粗比对湖羊瘤胃发酵和菌群结构的影响. 草业学报, 2020, 29(7): 122-130. doi: 10.11686/cyxb2019435 ZHAN J S, YANG Q, HU Y, WU Y P, HUO J H. Effects of concentrate to forage ratio on rumen fermentation and microbial community structure of Hu sheep. Acta Prataculturae Sinica, 2020, 29(7): 122-130. doi: 10.11686/cyxb2019435
[30] SPENCE C, WELLS W G, SMITH C J. Characterization of the primary starch utilization operon in the obligate anaerobe Bacteroides fragilis: Regulation by carbon source and oxygen. Journal of Bacteriology, 2006, 188(13): 4663-4672. doi: 10.1128/JB.00125-06
[31] MURO-REYES A, GUTIERREZ-BANUELOS H, DI-AZ-GARCI L H, GUTIERREZ F J, LUNA A C. Potential environmental benefits of residual feed intake as strategy to mitigate methaneemissions in sheep. Journal of Animal and Veterinary Advances, 2011, 10(12): 1551-1556. doi: 10.3923/javaa.2011.1551.1556
[32] NEGUSSIE E, MNTYSAARI, E A, LIADUER M H. Animal wise variation in enteric methane output traits and its relationship with feed efficiency in dairy cattle: A longitudinal model analysis. //10th World Congress on Genetics Applied to Livestock Production. Moscow: Pleiades Publishing, 2014: 85-91.
[33] 和东迁, 陶金忠. 反刍动物剩余采食量在瘤胃功能上的差异和相关分子生物学机制. 动物营养学报, 2021, 33(6): 3125-3131. HE D Q, TAO J Z. Differences in ruminant residual feed intake in rumen function and related molecular biological mechanisms. Chinese Journal of Animal Nutrition, 2021, 33(6): 3125-3131.
[34] ELLISON M J, CONANT G C, LAMBERSON W R, COCKRUM R R. AUSTINA K J, RULE C, CAMMACK K M. Diet and feed efficiency status affect rumen microbial profiles of sheep. Small Ruminant Re-search, 2017, 156: 12-19. doi: 10.1016/j.smallrumres.2017.08.009
[35] ZHOU M, HERNANDESANABRIA E, GUAN L L. Characterization of variation in rumen methanogenic communities under different dietary and host feed efficiency conditions, as determined by PCR-denaturing gradient gel electrophoresis analysis. Applied and Environmental Microbiology, 2010, 76(12): 3776-3786. doi: 10.1128/AEM.00010-10
[36] 董利锋, 付敏, 陈天宝, 刁其玉. 反刍动物瘤胃优势产甲烷菌菌群结构及多样性研究进展. 动物营养学报, 2019, 31(9): 3927-3935. DONG L F, FU M, CHEN T B, DIAO Q Y. Advances in the structure and diversity of dominant methanogens in rumen of ruminants. Chinese Journal of Animal Nutrition, 2019, 31(9): 3927-3935.
-
图 1 不同RFI组OTUsVenn图
EHF、EHM、ELF和ELM分别表示极高组母羊、极高组公羊、极低组母羊、极低组公羊;下同。
Figure 1. OTUs Venn diagram of different RFI groups
EHF, EHM, ELF, and ELM indicate extremely high groups of females and males, and low groups of females and males, respectively. This is applicable for the following tables and figures as well.
表 1 基础日粮组成及营养水平
Table 1 Composition and nutrient levels of basal diets
项目 Item 含量 Content 原料 Ingredient/% 玉米秸秆 Corn straw 20.0 玉米 Corn 32.0 糖蜜 Molasses 4.0 豆粕 Soybean meal 6.0 棉粕 Cotton meal 8.0 玉米皮 Corn bran 15.3 玉米胚芽粕 Corn germ meal 11.0 石粉 Limestone 1.2 食盐 NaCl 0.5 预混料 Premix 2.0 合计 Total 100.0 营养水平(干物质基础)
Nutrient level (Dry matter basis)1)代谢能 Metabolic energy/(MJ·kg−1) 9.93 粗蛋白质 Crude protein/% 13.74 粗脂肪 Ether extract/% 2.74 中性洗涤纤维 Neutral detergent fibers/% 35.99 酸性洗涤纤维 Acid detergent fibers/% 20.34 钙 Calcium/% 0.71 磷 Phosphorus/% 0.31 1)预混料为每千克饲粮提供:铁69.63 mg,铜7.41 mg,锰23.7 mg,锌55 mg,碘0.67 mg,钴0.3 mg,维生素A 2500 IU,维生素E 23 IU。2)代谢能为计算值,其他营养水平为测定值。
1)Premix provided the following per kilogram of the diet: Fe 69.63 mg, Cu 7.41 mg, Mn 23.7 mg, Zn 55 mg, I 0.67 mg, Co 0.3 mg, VA 2 500 IU, and VE 23 IU. 2)Metabolic energy calculated value, nutrient levels were measured values.表 2 不同剩余采食量对滩羊瘤胃微生物多样性影响
Table 2 Different residual feed intake on rumen microbial diversity of Tan sheep
项目
Item组别 Group P EHM ELM EHF ELF 观测到的物种数 No. of observed species 860.00 ± 27.37 838.00 ± 41.78 857.00 ± 68.35 837.00 ± 55.81 0.983 Shannon-Wiener 5.79 ± 0.35 6.33 ± 0.14 6.02 ± 0.49 6.01 ± 0.32 0.724 Simpson 0.91 ± 0.22 0.96 ± 0.01 0.94 ± 0.21 0.94 ± 0.14 0.265 Chao1 961.04 ± 30.71 926.89 ± 39.40 961.99 ± 65.24 937.14 ± 58.35 0.947 ACE 975.04 ± 29.93 947.22 ± 39.42 984.36 ± 67.55 962.56 ± 56.61 0.956 覆盖率 Good’s_coverage 0.996 0 ± 0.000 4 0.996 0 ± 0.000 1 0.996 0 ± 0.000 3 0.996 0 ± 0.000 2 0.573 PD_whole_tree 71.87 ± 2.33b 102.23 ± 11.78a 74.92 ± 7.73b 74.44 ± 6.86 0.058b 同行不同大小写字母分别表示在0.05和0.1水平上差异显著。下表同。
Different capital and lowercase letters indicate significant differences at the 0.05 and 0.1 levels, respectively. this is appicable for the following tables as well.表 3 微生物相对丰度在门分类水平上排名前十的物种
Table 3 Top 10 species at phylum taxonomic level in relative abundance of microorganisms
门
Phlyum组别 Group P EHM ELM EHF ELF 拟杆菌门 Bacteroidota 49.58 ± 5.56A 36.66 ± 3.38B 27.48 ± 4.42B 33.67 ± 4.57B 0.022 放线菌门 Actinobacteriota 3.51 ± 0.81c 7.05 ± 3.30b 27.10 ± 8.90a 16.15 ± 7.12ab 0.057 厚壁菌门 Firmicutes 38.08 ± 6.35 41.47 ± 4.16 36.11 ± 5.05 42.27 ± 5.41 0.811 广古菌门 Euryarchaeota 2.81 ± 1.01B 7.95 ± 1.62A 5.55 ± 0.73A 4.88 ± 0.95A 0.050 变形菌门 Proteobacteria 2.80 ± 1.07A 0.99 ± 0.26B 0.70 ± 0.18B 0.54 ± 0.13B 0.019 未分类 Unidentified_Bacteria 0.88 ± 0.13 1.65 ± 0.33 1.27 ± 0.37 0.73 ± 0.12 0.115 螺旋菌门 Spirochaetota 0.15 ± 0.05 0.43 ± 0.14 0.14 ± 0.05 0.17 ± 0.08 0.115 蓝藻门 Cyanobacteria 0.15 ± 0.06 0.24 ± 0.10 0.18 ± 0.08 0.19 ± 0.06 0.592 纤维杆菌门 Fibrobacterota 0.11 ± 0.07 0.11 ± 0.03 0.08 ± 0.05 0.04 ± 0.02 0.631 酸杆菌门 Acidobacteriota 0.06 ± 0.02 0.05 ± 0.02 0.09 ± 0.03 0.08 ± 0.04 0.426 表 4 微生物相对丰度在属分类水平上排名前十的物种
Table 4 Top ten species at genus taxonomic level in relative abundance of microorganisms
属
Genus组别 Group P EHM ELM EHF ELF Olsenella 2.89 ± 0.80b 4.66 ± 2.06b 23.66 ± 8.84a 13.84 ± 6.80a 0.069 普雷沃氏菌属 Prevotella 32.66 ± 5.05a 20.44 ± 4.09ab 16.00 ± 4.00b 16.83 ± 9.23b 0.057 韦荣球菌属 UCG_002 Erysipelotrichaceae_UCG_002 14.22 ± 7.41A 0.26 ± 0.05B 3.37 ± 2.40B 0.10 ± 0.02B 0.028 Quinella 0.02 ± 0.00 1.73 ± 1.65 0.42 ± 0.10 5.10 ± 4.76 0.522 月形单胞菌属 Selenomonas 3.91 ± 1.79ab 8.21 ± 2.80a 1.73 ± 0.43b 2.68 ± 1.27b 0.094 甲烷短杆菌 Methanobrevibacter 2.77 ± 0.10b 7.82 ± 1.62a 5.51 ± 0.72b 4.81 ± 0.95b 0.055 双歧杆菌属 Bifidobacterium 0.34 ± 0.05 2.06 ± 1.50 2.84 ± 1.31 1.73 ± 0.52 0.531 Acetitomaculum 1.58 ± 0.25 2.96 ± 1.38 2.93 ± 1.09 3.79 ± 1.12 0.634 瘤胃球菌属 Ruminococcus 2.13 ± 0.77 4.12 ± 1.29 1.69 ± 0.43 2.14 ± 0.82 0.216 毛螺菌 NK3A20 Lachnospiraceae_NK3A20_group 1.85 ± 1.24 3.90 ± 1.01 3.28 ± 0.68 3.36 ± 0.66 0.420 -
[1] KOCH R M, SWIGER L A, CHAMBERS D, GREGORY K E. Efficiency of feed use in beef cattle. Journal of Animal Science, 1963, 22(2): 486-494. doi: 10.2527/jas1963.222486x
[2] HERD R M, ARTHUR P F. Physiological basis for residual feed intake. Journal of Animal Science, 2009, 87 (14 Suppl): 64-71.
[3] AHMED A, ELOLIMY, MOHAMED K, ABDELMEGEI D, JOSHUA C, MCCANN, DANIL W, SHIKE, JUAN J L. Residual feed intake in beef cattle and its association with carcass traits, ruminal solid-fraction bacteria, and epithelium gene expression. Journal of Animal Science and Biotechnology, 2018(9): 67.
[4] PACHECO D, WAGHORN G, JANSSEN P H. Decreasing methane emissions from ruminants grazing forages: A fit with productive and financial realities. Animal Production Science, 2014, 54(9): 1141-1154. doi: 10.1071/AN14437
[5] GUAN L L, NRUMAH J D, BASARAB J A, MOORE S S. Linkage of microbial ecology to phenotype: Correlation of rumen microbial ecology to cattle’s feed efficiency. FEMS Microbiology Letters, 2008, 288(1): 85-91. doi: 10.1111/j.1574-6968.2008.01343.x
[6] DÉBORAH R G L, MÁRCIO D S D, ALEX J L R. Assessing the relationship between the rumen microbiota and feed efficiency in Nellore steers. Journal of Animal Science and Biotechnology, 2021, 12(4): 1645-1661.
[7] ZHANG Y K, ZHANG X X, LI F D, LI C, ZHANG D Y, SONG Q Z, LI X L, ZHAO Y, WANG W M. Characterization of the rumen microbiota and its relationship with residual feed intake in sheep. Animal, 2021, 15(3): 100161. doi: 10.1016/j.animal.2020.100161
[8] ELLISON M J, CONANT G C, LAMBERSON W R, COCKRUM R R, AUSTIN K J, RULE D C, CAMMACK K M. Diet and feed efficiency status affect rumen microbial profiles of sheep. Small Ruminant Research, 2017, 156(11): 12-19.
[9] CLEMONS B A, SHIN S B, SMITH T P L, EMBREE M M, VOY B H, SCHNEIDER L G, DONOHOE D R, MCLEAN Kyle J, MYER P R. Ruminal protozoal populations of angus steers siffering in feed efficiency. Animals, 2021, 11(6): 1561. doi: 10.3390/ani11061561
[10] LIANG Y, LI G, LI X, LU J, LI F, TANG D. Growth performance, rumen fermentation, bacteria composition, and gene expressions involved in intracellular ph regulation of rumen epithelium in finishing Hu lambs differing in residual feed intake phenotype. Journal of Animal Science, 2017, 95(4): 1727-1738.
[11] 陶金忠, 和东迁, 王俊奎, 陈丽尧, 罗芳, 张倩, 卢童童, 吴少飞. 一种滩羊单栏饲喂设备: ZL 213603732U, 2021-07-06. TAO J Z, HE D Q, WANG J K, CHEN L Y, LUO F, ZHANG Q, LU T T, WU S F. The utility model relates to a single stall feeding device for Tan sheep: ZL 213603732U, 2021-07-06.
[12] 中华人民共和国农业部. NY/T816–2004 肉羊饲养标准. 北京: 中国标准出版社, 2004. Ministry of Agriculture of the People's Republic of China. NY/T816–2004 Feeding Standad of Meat-Producing Sheep and Goats. Beijing: China Standard Press, 2004.
[13] 陈丽尧, 和东迁, 卢童童, 张倩, 吴少飞, 罗芳, 王俊奎, 张瑞雪, 陶金忠. 在不限定运动的情况下滩羊RFI的测定及与其他生长性能之间的关系. 中国兽医学报, 2022, 42(1): 2056-2061. CHEN L Y, HE D Q, LU T T, ZHANG Q, WU S F, LUO F, WANG J K, ZHANG R , TAO J Z. Masurement of RFI traits of Tan sheep and the relationship with other growth perfprmance without restricting exercise. Chinese Journal Veterinay Science, 2022, 42(1): 2056-2061.
[14] ZHANG X X, LI G Z, LI F D, ZHANG D Y, YUAN L F, ZHAO Y, ZHANG Y K, LI X L, SONG Q Z, WANG W M. Effect of feed efficiency on growth performance, body composition, and fat deposition in growing Hu lambs. Animal Biotechnology, 2021(11): 1-16.
[15] CHEN L Y, HE D Q, LU T T, ZHANG Q, WU S F, LUO F, WANG J K, ZHANG R X, TAO J Z. Measurement of RFI of Tan sheep without restricted exercise and its relationship with other growth performance. Chinese, 2022, 42(1): 6.
[16] LI F, LE L G. Metatranscriptomic profiling reveals linkages between the active rumen microbiome and feed efficiency in beef cattle. Applied and Environmental Microbiology, 2017, 83(9): e00061-17.
[17] 石婧, 姚勤, 陈克平. 性别对生物生长发育速率影响的研究. 安徽农业科学, 2011, 39(22): 13301-13302, 13305. doi: 10.3969/j.issn.0517-6611.2011.22.018 SHI J, YAO Q, CHEN K P. Effects of sex on biological growth and development rate. Journal of Anhui Agricultural Sciences, 2011, 39(22): 13301-13302, 13305. doi: 10.3969/j.issn.0517-6611.2011.22.018
[18] VERSCHUREN L M G, CALUS M P L, JANSMAN A J M, BERGSMA R, KNOL E F, GILBERT H, ZEMB O. Fecal microbial composition associated with variation in feed efficiency in pigs depends on diet and sex. Journal of Animal Science, 2018, 96(9): 1405-1418.
[19] 韩学平, 刘宏金, 胡林勇, 徐世晓, 艾德强, 付弘贇, 陈永伟, 赵建忠. 环湖牦牛瘤胃微生物区系特征及性别之间的差异. 动物营养学报, 2020, 32(1): 234-243. doi: 10.3969/j.issn.1006-267x.2020.01.029 HAN X P, LIU H J, HU L Y, XU S X, AI D Q, FU H Y, CHEN Y W, ZHAO J Z. Characteristics and gender differences of rumen microflora in yaks around lake. Chinese Journal of Animal Nutrition, 2020, 32(1): 234-243. doi: 10.3969/j.issn.1006-267x.2020.01.029
[20] ELIE J, WHITE B A, ITZHAK M, HEIMESAAT M M. Potential role of the bovine rumen microbiome in modulating milk composition and feed efficiency. PLoS One, 2014, 9(1): e85423. doi: 10.1371/journal.pone.0085423
[21] PATIL R D, ELLISON M J, WOLFF S M, SHEARER, COURTNEY, WRIGHT A, COCKRUM R, AUSTIN K, LAMBERSON W, CAMMACK K, CONANT G. Poor feed efficiency in sheep is associated with several structural abnormalities in the community metabolic network of their ruminal microbes. Journal of Animal Science, 2018, 96(6): 2113-2124. doi: 10.1093/jas/sky096
[22] 马万浩, 梁玉生, 张智安, 董春晓, 李飞, 李发弟, 翁秀秀. 不同剩余采食量的湖羊生长性能和消化道微生物多样性差异的研究. 动物营养学报, 2019, 31(9): 4080-4091. MA W H, LIANG Y S, ZHANG Z A, DONG C X, LI F, LI F D, WENG X X. Difference of grobial diversity of Hu sheep with different residual feed intake. Chinese Journal of Animal Ntrition, 2019, 31(9): 4080-4091.
[23] MYER P R, SMITH T, WELLS J E, KUEHN L A, FREETLY H C, FORSTER R J. Rumen microbiome from steers differing in feed efficiency. PLoS One, 2015, 10(6): e0129174. doi: 10.1371/journal.pone.0129174
[24] WANG W, LI C, LI F, WANG X, ZHANG X, LIU T. Effects of early feeding on the host rumen transcriptome and bacterial diversity in lambs. Scientific Reports, 2016, 6(8): 32479.
[25] LI F, LI C, CHEN Y, MALLORY M E, BRYNN H V, LIESEL G S, DALLAS R D, KYLE J M, PHILIIP R M. Host genetics influence the rumen microbiota and heritable rumen microbial features associate with feed efficiency in cattle. Microbiome, 2019, 7(1): 92. doi: 10.1186/s40168-019-0699-1
[26] 张小雪. 不同剩余采食量羔羊生产性能和瘤胃微生物区系及肝脏转录组研究. 兰州: 兰州大学博士学位论文, 2019. ZHANG X X. Study on performance, rumen microflora and liver transcriptome of lambs with different residual feed intake. PhD Thesis. Lanzhou: Lanzhou University, 2019.
[27] 孙纪录, 贾英民, 高小芹, 田洪涛, 张伟. 菊芋在体内对双歧杆菌生长的影响研究. 食品科技, 2007, 32(1): 212-214. doi: 10.3969/j.issn.1005-9989.2007.01.064 SUN J L, JIA Y M, GAO X Q, TIAN H S, ZHANG W. Effects of Jerusalem artichos on the growth of bifidobacteria in vivo. Food Science and Technology, 2007, 32(1): 212-214. doi: 10.3969/j.issn.1005-9989.2007.01.064
[28] PEREA K, PERZ K, OLIVO S K, WILLIAMS A, YEOMAN C J. Feed efficiency phenotypes involve changes in ruminal, colonic, and small intestine-located microbiota. Journal of Animal Science, 2017, 95(6): 2585-2592.
[29] 占今舜, 杨群, 胡耀, 武艳平, 霍俊宏. 日粮精粗比对湖羊瘤胃发酵和菌群结构的影响. 草业学报, 2020, 29(7): 122-130. doi: 10.11686/cyxb2019435 ZHAN J S, YANG Q, HU Y, WU Y P, HUO J H. Effects of concentrate to forage ratio on rumen fermentation and microbial community structure of Hu sheep. Acta Prataculturae Sinica, 2020, 29(7): 122-130. doi: 10.11686/cyxb2019435
[30] SPENCE C, WELLS W G, SMITH C J. Characterization of the primary starch utilization operon in the obligate anaerobe Bacteroides fragilis: Regulation by carbon source and oxygen. Journal of Bacteriology, 2006, 188(13): 4663-4672. doi: 10.1128/JB.00125-06
[31] MURO-REYES A, GUTIERREZ-BANUELOS H, DI-AZ-GARCI L H, GUTIERREZ F J, LUNA A C. Potential environmental benefits of residual feed intake as strategy to mitigate methaneemissions in sheep. Journal of Animal and Veterinary Advances, 2011, 10(12): 1551-1556. doi: 10.3923/javaa.2011.1551.1556
[32] NEGUSSIE E, MNTYSAARI, E A, LIADUER M H. Animal wise variation in enteric methane output traits and its relationship with feed efficiency in dairy cattle: A longitudinal model analysis. //10th World Congress on Genetics Applied to Livestock Production. Moscow: Pleiades Publishing, 2014: 85-91.
[33] 和东迁, 陶金忠. 反刍动物剩余采食量在瘤胃功能上的差异和相关分子生物学机制. 动物营养学报, 2021, 33(6): 3125-3131. HE D Q, TAO J Z. Differences in ruminant residual feed intake in rumen function and related molecular biological mechanisms. Chinese Journal of Animal Nutrition, 2021, 33(6): 3125-3131.
[34] ELLISON M J, CONANT G C, LAMBERSON W R, COCKRUM R R. AUSTINA K J, RULE C, CAMMACK K M. Diet and feed efficiency status affect rumen microbial profiles of sheep. Small Ruminant Re-search, 2017, 156: 12-19. doi: 10.1016/j.smallrumres.2017.08.009
[35] ZHOU M, HERNANDESANABRIA E, GUAN L L. Characterization of variation in rumen methanogenic communities under different dietary and host feed efficiency conditions, as determined by PCR-denaturing gradient gel electrophoresis analysis. Applied and Environmental Microbiology, 2010, 76(12): 3776-3786. doi: 10.1128/AEM.00010-10
[36] 董利锋, 付敏, 陈天宝, 刁其玉. 反刍动物瘤胃优势产甲烷菌菌群结构及多样性研究进展. 动物营养学报, 2019, 31(9): 3927-3935. DONG L F, FU M, CHEN T B, DIAO Q Y. Advances in the structure and diversity of dominant methanogens in rumen of ruminants. Chinese Journal of Animal Nutrition, 2019, 31(9): 3927-3935.
-
期刊类型引用(9)
1. 万建琦,丁瑜,任晗,李雯霞,杨勇,黄金盟,关长飞. DdSOC1基因调控德阳柿成花分子机制. 果树学报. 2025(02): 276-287 .  百度学术
百度学术
2. 杨胤延,孙东宇,蔡昕成,胡志群,吴国麟,周碧燕. 莲雾调控开花基因SsSOC1的克隆及表达分析. 热带作物学报. 2025(03): 553-562 .  百度学术
百度学术
3. 龙丽君,马英姿,曾慧杰,李昌珠,张岗,刘思思,李依民. 灰毡毛忍冬花器官发育相关LmSOC1基因克隆及表达分析. 中南林业科技大学学报. 2024(05): 181-190 .  百度学术
百度学术
4. 俞亚欣,龙丽君,李昌珠,曾慧杰,乔中全,刘思思,马英姿. 灰毡毛忍冬AP1同源基因的克隆及互作蛋白鉴定. 药学学报. 2024(10): 2880-2888 .  百度学术
百度学术
5. 梁婷,阙远慧,喻奇伟,贾傛吏,邹广,郭凯阳,黄莺,刘仁祥. 烤烟品种'韭菜坪2号'叶片数的遗传分析及基因定位. 农业生物技术学报. 2023(01): 50-60 .  百度学术
百度学术
6. 崔秀文,刘迪,黄天苗,李美玲,栗孟飞,魏建和. 当归MADS-box生物信息学及SOC1克隆与表达分析. 中草药. 2023(05): 1551-1560 .  百度学术
百度学术
7. 蔺悦,陈俊兴,红梅,陈一潇,徐宛玉,张雅茜,包福海,乌云塔娜,王淋,包文泉. 西伯利亚杏PsSOC1-like基因密码子偏好性与进化分析. 内蒙古农业大学学报(自然科学版). 2023(01): 13-25 .  百度学术
百度学术
8. 刘朝斌,李荣,陈诗婷,张茜,张振源. 核桃开花相关基因JrSOC1的筛选及其在开花中的作用分析. 西北林学院学报. 2023(04): 97-103 .  百度学术
百度学术
9. 张丽杰,董天一,吴静雯,张萌萌,贾若雪,刘春平. 胡桃楸成花相关基因JmSOC1克隆和表达分析. 沈阳农业大学学报. 2023(06): 673-682 .  百度学术
百度学术
其他类型引用(11)
-
其他相关附件



 下载:
下载: