第一作者:李永青(1992-),女,甘肃渭源人,在读硕士生,主要从事动物营养与饲料科学研究。E-mail:m18894036031@163.com
DNA条形码技术是利用DNA保守片段对物种进行快速准确鉴定的新兴技术。本研究根据GenBank中禾本科牧草 matK和 rbcL基因的核苷酸序列,设计4对通用引物,建立并优化了针对禾本科7个主要牧草属8种牧草11个样品[高丹草( Sorghum bicolor×S. sudanense)、玉米( Zea mays)、针茅( Stipa capillata)、“贝克”多年生黑麦草( Lolium perenne ‘Plxie)、“凯帝莎”多年生黑麦草( L. perenne ‘Caddieshack’)、甘肃羊茅( Festuca kansuensis)、“百琪”紫羊茅( F. rubra ‘Bargena’)、“梦神”紫羊茅( F. rubra ‘Maxima’)、“百胜”草地早熟禾( Poa pratensis ‘Barvictor’)、“钻石”草地早熟禾( P. pratensis ‘Diamond’)和芨芨草( Achnatherum splendens)]的目的片段的扩增条件。对扩增产物进行测序和分析,经分别比对,筛选出8种牧草的4个标记位点5’端和3’端保守序列。对各标记位点保守区内的核苷酸进行单核苷酸多态性(SNPs)的单倍型分析。结果表明, matK1、 matK2、 matK3分别有6个单倍型(H1A、H1B、H1C、H1D、H1E和H1F)、7个单倍型(H2A、H2B、H2C、H2D、H2E、H2F和H2G)和3个单倍型(H3A、H3B和H3C), rbcL基因有5个单倍型(H4A、H4B、H4C、H4D和H4E)。根据 matK( matK1、 matK2、 matK3)和 rbcL基因筛选的4个标记位点为8种牧草建立了相对应的特异DNA识别码。本研究可为混合禾本科牧草饲料中的高粱属、玉蜀黍属、芨芨草属、针茅属、黑麦草属、羊茅属和早熟禾属的8种牧草准确识别提供分子水平上的科学依据。
DNA barcoding is an emerging technology, which is used as a tool to indentify species accurately and quickly using the conservative DNA segments. Our research has established and optimized target fragments’ the amplification conditions for eleven samples ( Sorghum bicolor×Sorghum sudanense, Zea mays, Stipa capillata, Lolium perenne ‘Plxie’, Lolium perenne ‘Caddieshack’, Festuca kansuensis, Festuca rubra ‘Bargena’, F. rubra ‘Maxima’, Poa pratensis ‘Barvictor’, P. pratensis ‘Diamond’, Achnatherum splendens) from seven major genera ( Sorghum, Zea, Stipa, Lolium, Festuca, Poa, Achnatherum) and eight species according to nucleotide sequence of grass matK and rbcL genes in GenBank, and designed four universal primers. 5' end and 3' end conservative fragments in four marked fragments were selected by amplification products sequencing and analyzing. Single nucleotide polymorphism (SNP) haplotype analysis of conservative fragments nucleotide sequences of each maker loci showed there were 6 haplotypes(H1A, H1B, H1C, H1D, H1E and H1F), 7 haplotypes (H2A, H2B, H2C, H2D, H2E, H2F and H2G) and 3 (H3A, H3B and H3C) haplotypes among matK1, matK2, matK3 respectively, by the same token, rbcL gene has 5 haplotypes (H4A, H4B, H4C, H4D and H4E). According to the haplotypes about matK (matK1, matK2 and matK3) and rbcL, we have constructed the DNA barcoding for eight grass species. The research results provide a scientific basis for identification of mixed feed grasses including eight species of the seven generas.
DNA条形码技术又称DNA条形编码, 因其快速、简便、精准等特点常被用作物种鉴定, 其原理是基于物种种内特异性和种间多样性利用一个或几个标准的DNA片段(DNA barcode)构建生物鉴别数据库[1, 2]。Hebert等[3]在对11种不同门的动物的研究中发现, COI(cytochrome coxi-dase subunit 1)基因序列的种间变异能够较好地区分物种, 而植物中的COI或其它线粒体基因的变异度较低, 不适用于植物物种的鉴定。据报道, 植物中的叶绿体基因虽相对保守, 但其单亲遗传避免了基因重组, 且含量丰富, 扩增能力强, 故在叶绿体基础上进行植物物种的鉴定成为一种可行的办法。国际生命条形码联盟植物工作组[4]建议将rbcL+matK组合作为陆地植物的核心DNA条形码, 用于构建植物物种鉴定的统一框图。我国植物条形码研究团队基于更大规模取样的比较分析, 提出了将ITS或ITS2纳入种子植物核心条形码的建议[5, 6]。Costion等[7]利用谱系多样性指数识别热带雨林避难所和物种分化中心以确定生物多样性保护重点。基于ITS2序列, 可以快速并有效地从豆蔻属(Amomum)和山姜属(Alpinia)中鉴别出中药材[8]。
由于植物条形码目前处于对片段的研究阶段, 其分析方法与动物有所不同。首先进行序列比对和人工校正, 删除位于序列两端的不可靠碱基, 利用PAUP或MEGA软件, 采用pairwise uncorrected p-distance[9]或Kimura-2-parameter distance(K2P)模型[10, 11]计算种内和种间的遗传距离; 采用分子系统学方法, 通过构建多种系统发育树对条形码进行分析(如NJ、UPGMA、ML和MP等)并检验每个物种的单系情况。
禾本科牧草作为家畜日粮的主要饲草成分[12], 分布较广, 饲用意义较大的包括30属[13], 大多为牲畜所喜食。然而DNA条形码技术在牧草鉴别中应用较为罕见, 本研究借助分子遗传学方法, 尝试建立禾本科7个属8种牧草的DNA条码, 旨为进一步开展品种鉴别提供理论依据和方法参考。
1.1.1 样品 本研究共采集到禾本科牧草7个属8个种共11个样品, 样品信息详见表1。
| 表1 本研究所用样品信息 Table 1 Information of samples in this study |
1.1.2 PCR引物设计 参照GenBank(http://www.ncbi.nlm.nih.gov)中叶绿体matK和rbcL基因的核苷酸序列, 采用Primers 5.0 (http://www.bbioo.com/download/58-166-1.html) 进行引物设计。其中, matK基因参照高粱属(Sorghum)(AF164418、HF558520)、羊茅属(Festuca)(DQ786940)、燕麦属(Avena)(GU367310、GU367311)、大麦属(Hordeum)(AB078131、AB078133)、稻属(Oryza)(AF489915、AY768779)和针茅属(Stipa)(KC129652、KC129653)设计3对引物; rbcL基因参照芨芨草属(Achnatherum)(HQ600430)、针茅属(HE573441)、高粱属(HQ600085)和羊茅属(FN668434、FN668435)设计1对引物(表2)。引物由上海生工生物工程有限公司合成。
| 表2 引物PCR反应最佳退火温度表 Table 2 The optimized annealing temperature of primer’ s PCR reaction |
1.2.1 基因组DNA提取、PCR扩增和测序 称取植物叶片约10 mg, 用液氮研磨至粉末后, 采用CTAB法提取基因组DNA[14], TE溶解。PCR扩增采用50 μ L体系:dNTP(2.5 mmol· L-1)2 μ L, 10× Buffer 5 μ L, Taq DNA 聚合酶(5 U· μ L-1)0.4 μ L, DNA 模板2 μ L, 上、下游引物各1 μ L(10 μ mol· L-1), 加ddH2O至50 μ L。PCR反应条件:94 ℃预变性2min; 94 ℃变性40 s, 退火30 s、72 ℃延伸30 s(30个循环); 72 ℃延伸10 min; 4 ℃保存。PCR扩增产物经琼脂糖凝胶电泳(30 min, 150 V)检验后送上海生工生物工程有限公司进行测序。
1.2.2 数据处理 利用chromas 2.33(http://www.seekbio.com/DownloadShow.asp?id=284)对测序结果进行校对和编辑, 并利用MEGA 5.0(http://mega.software.informer.com/5.0/)进行序列比对, 同时借助Dnasp 5.10(http://www.itopdog.cn/soft/4785.html)分析单倍型和变异位点。
根据本研究设计的4对通用引物, 并针对禾本科11个牧草样品建立并优化目的片段PCR条件。
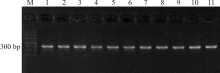
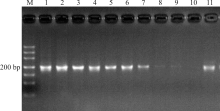
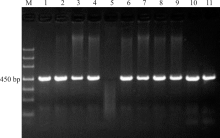
上述电泳检测结果获得的目的条带清晰(图1-4), 片段大小与预期结果相符, 经测序获得较高质量的核苷酸序列, 说明本研究筛选的引物可用于DNA条码的筛选。
 | 图1 引物F-M1/R-M433 PCR扩增凝胶电泳检测图 注:M, Marker DL1000; 1, 高丹草; 2, 玉米; 3, 针茅; 4, “ 贝克” 多年生黑麦草; 5, “ 凯帝莎” 多年生黑麦草; 6, 甘肃羊茅; 7, “ 百琪” 紫羊茅; 8, “ 梦神” 紫羊茅; 9, “ 百胜” 草地早熟禾; 10, “ 钻石” 草地早熟禾; 11, 芨芨草。图2同。Fig.1 Detection of primer F-M1/R-M433 PCR products in gel electrophoresis Note: M, Marker DL1000; 1, Sorghum bicolor× Sorghum sudanense; 2, Zea mays; 3, Stipa capillata; 4, Lolium perenne ‘ Plxie’ ; 5, Lolium perenne ‘ Caddieshack’ ; 6, Festuca kansuensis; 7, Festuca rubra ‘ Bargena’ ; 8, Festuca rubra ‘ Maxima’ ; 9, Poa pratensis ‘ Barvictor’ ; 10, Poa pratensis ‘ Diamond’ ; 11, Achnatherum splendens. The same in Fig.2. |
利用chromas 2.33对测序结果进行校对和编辑后, 通过MEGA 5.0进行序列比对, 分别筛选标记位点5’ 端和3’ 端高度保守区(表3)。
| 表3 标记位点保守区识别 Table 3 Identification of conserved region in fragment marked |
2.3.1 matK1 基于Dnasp 5.10进行单倍型分析, 8种牧草11个样品彼此间有24个变异位点, 分为6个单倍型(表4)。其中, H1A、H1B、H1D、H1E和H1F分别为高丹草、针茅、甘肃羊茅、多年生黑麦草和紫羊茅的特有单倍型; H1C为草地早熟禾、玉米和芨芨草的共享单倍型。另外, 同一种牧草的不同品种间均具有共享单倍型(表4)。
| 表4 matK1单倍型多态位点 Table 4 matK1 haplotypes polymorphic sites |
2.3.2 matK2 8种牧草11个样品彼此间有8个变异位点, 分为7个单倍型(表5)。H2A、H2C、H2D、H2E、H2F和H2G分别为玉米、针茅、高丹草、甘肃羊茅、多年生黑麦草和紫羊茅6种牧草的特有单倍型; H2B为牧草芨芨草和草地早熟禾的共享单倍型。另外, 同一种牧草的不同品种间均具有共享单倍型。
| 表5 matK2单倍型多态位点 Table 5 matK2 haplotypes polymorphic sites |
2.3.3 matK3 在matK3片段上设计的引物未能成功扩增出多年生黑麦草的两个品种贝克和凯帝莎, 扩增得到9个样品彼此间有5个变异位点, 分为3个单倍型(表6)。其中, 仅针茅有其特有单倍型H3B; H3A分别为甘肃羊茅、芨芨草、草地早熟禾和紫羊茅4种牧草的共享单倍型; H3C为高丹草和玉米的共享单倍型。
| 表6 matK3单倍型多态位点 Table 6 matK3 haplotypes polymorphic sites |
2.3.4 rbcL序列 在rbcL片段上设计的引物扩增所得到的10个样品中有33个变异位点, 分为5个单倍型(表7)。其中, H4B、H4C和H4D分别为草地早熟禾、针茅和多年生黑麦草3种牧草的特有单倍型; H4A为高丹草和玉米的共享单倍型; H4E为紫羊茅和甘肃羊茅的共享单倍型。
| 表7 rbcL单倍型多态位点 Table 7 rbcL haplotypes polymorphic sites |
基于matK 3个标记位点(matK1、matK2、matK3)和rbcL基因在牧草种属间表达的特异性建立了8种牧草的DNA条形码数据库, 该数据库由matK 3个标记位点和rbcL基因的特异性引物(表2)、标记位点5’ 端和3’ 端保守序列(表3)以及DNA识别码(表8)共同组成。同一种牧草的不同品种, 如草地早熟禾两个品种钻石和百胜具有共同的DNA barcoding, 与其它属的牧草DNA barcoding完全不同, 验证了DNA条形码种内无差异、种间允许有较大差异的特征, 达到了鉴别的标准。
| 表8 8种牧草的DNA Barcoding 数据库 Table 8 The Databas of DNA Barcoding for 8 species pasture |
生物DNA 条形码联盟(consortium for the barcode of life, CBOL)提议的植物DNA条形码候选片段有matK、rpoB、rpoC1、rbcL和psbA-trnH等序列, 其中rbcL、matK和ITS为核心条形码[4], trnH-psbA为补充条形码[16]。有研究报道, matK具有进化速率快, 种间鉴别能力较高等特点, 但引物通用性差[17]。葡萄科崖爬藤属的matK扩增成功率达到93.5%, 扩增效果很理想, 但单独使用时物种识别率不高, 仅为22.2%[18]。rbcL序列具有通用性高、易扩增、易比对等特点, 但变异主要存在于种以上水平, 物种水平上通常变异不够大[19, 20, 21]。尽管rbcL不能识别全部物种, 但可以区分不少同属植物[22]。因此, 几位研究者都曾建议将rbcL与另外一个或多个片段组合使用[18, 22]。
本研究选择matK和rbcL基因进行研究, 判定其是否符合DNA条形码的候选条件。高度保守的通用引物设计是理想的DNA barcoding序列获得的前提[23]。通过对GenBank中高粱属、燕麦属、大麦属、稻属、针茅属和芨芨草属等禾本科牧草叶绿体matK基因和rbcL基因的多重比对, 筛选出高度保守区域, 并进行引物设计。获得的matK (matK1、matK2、matK3)和rbcL 共4个标记位点的引物(表2), 实现了目的片段地成功扩增和测序, 结果较理想, 为DNA barcoding数据库的建立提供了可能性。DNA条形码的核心是PCR扩增, 而退火温度是影响PCR扩增的重要因素, 在Tm值允许范围内, 选择较高的复性温度可相对减少引物和模板间的非特异性结合, 提高PCR扩增的特异性[24]。电泳结果表明, 所设计引物扩增效率高、成功率高、反应条件适宜, 为物种的鉴别奠定了基础。
通过比较matK基因3个标记位点单倍型结果发现, matK基因3个标记位点(matK1、matK2、matK3)保守区片段大小依次较小, 变异位点数也依次降低。在属水平上, matK1无法区分早熟禾属、羊茅属和芨芨草属, 对于其余4个属的牧草能准确鉴别, 属水平的鉴别成功率为57.1%, 种水平的鉴别成功率为62.5%。matK2无法区分芨芨草属和早熟禾属, 属水平鉴别成功率为71.4%, 种水平鉴别成功率为 75%。而matK3扩增成功率较低, 未扩增出黑麦草的两个品种, 存在5个变异位点, 将8种牧草分为3个单倍型, 仅区分出针茅属, 属水平鉴别成功率仅为16.7%, 与种水平相同。
通过分析rbcL鉴定结果发现, 在rbcL标记位点的保守区内存在33个变异位点, 相比于matK基因的3个标记位点, rbcL变异最高, 分为5个单倍型(H4A、H4B、H4C、H4D和H4E), 其中H4A、H4B、H4C和H4D之间相同的变异位点较多, 而与甘肃羊茅和紫羊茅所属的单倍型H4E相比均存在较大的差异。单倍型结果分析表明, rbcL基因仅能鉴别出早熟禾属的草地早熟禾, 黑麦草属的多年生黑麦草和针茅属的针茅3种牧草, 其余5种牧草存在共享单倍型, 无法鉴别。该结果符合rbcL基因需与其它片段组合使用的结论[22]。
本研究选择matK和rbcL基因联合使用进行禾本科牧草的鉴别, 4个标记位点的单倍型组合构成DNA识别码, 每种牧草都有其特异的DNA识别码, 在属水平和种水平鉴别成功率均达到100%。而这种组合方式无法鉴别同一种牧草的不同品种, 例如草地早熟禾的两个品种钻石和百胜具有共同的DNA识别码, 此外, 多年生黑麦草的两个品种贝克和凯帝莎以及紫羊茅的两个品种百琪和梦神均出现上述结果。
本研究通过分析多态位点, 界定单倍型, 通过标记位点的不同单倍型组合实现8种牧草的鉴定, 同时利用K2P模型构建系统发育树, 8种牧草分列于不同的进化支。再次验证了matK和rbcL片段组合后, 其鉴别效果明显高于单片段。系统发育多样性不受物种分类地位变更的影响, 是生物多样性研究的一个里程碑式的指数[25, 26, 27]。李陈建等[28]对30份苏丹草种质资源的19个性状进行聚类分析, 明确了苏丹草种质资源的不同类型。本研究通过构建系统发育树, 充分证明了禾本科牧草各物种的单系性, 其结果满足DNA barcoding对包含足够系统进化信息进行物种在分类系统(科、属等) 定位中的要求[23]。
由上述结果可知, matK基因(matK1、matK2、matK3)和rbcL基因在种水平上的鉴定成功率较高。由于matK基因(matK1、matK2、matK3)和rbcL基因对部分物种仍然不能较好地区分, 两种片段组合后能将禾本科8种牧草完全区分, 鉴定效率达到100%。因此, 将matK与rcbL联合应用作为禾本科牧草的DNA条形码通用序列组合。本研究结果为混合禾本科牧草饲料中的高丹草、玉米、芨芨草、针茅、多年生黑麦草、甘肃羊茅、紫羊茅和草地早熟禾的准确识别提供了分子水平上的科学依据。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|