第一作者:陶晓丽(1990-),女,甘肃民勤人,在读硕士生,主要从事草坪生物学与生物技术研究。E-mail:taoxl14@lzu.edu.cn
箭筈豌豆( Vicia sativa)是自花授粉的二倍体一年生豆科牧草,可为我国高海拔地区的反刍动物提供优质蛋白粗饲料。以“兰箭3号”春箭筈豌豆为研究对象,采用DNase I法纯化叶绿体,利用第二代高通量测序平台Illumina Hiseq 2000进行测序,并对“兰箭3号”叶绿体全基因组序列的结构特征进行分析。结果表明,“兰箭3号”叶绿体基因组仅包括一个单拷贝的反向重复序列,其叶绿体全基因组大小为121 883 bp,共编码了109个基因,包括4个核糖体RNA(rRNA)基因,29个转运RNA(tRNA)基因,75个蛋白质编码基因和1个假基因。“兰箭3号”在叶绿体基因组结构、基因种类、排列顺序上与其它豆科植物基本一致。其叶绿体全基因组序列已在GenBank注册,序列号为KU053796。“兰箭3号”叶绿体全基因组测序的成功为叶绿体分子生物学研究奠定了基础,并可有效地促进箭筈豌豆遗传育种和分子进化研究。
The common vetch ( Vicia sativa) is a self-pollinating and diploid annual legume, which provides high-quality protein roughage for ruminants of high altitude areas in China. In this study, we evaluated the V. sativa cv. Lanjian No.3 using Illumina Hiseq 2000 to analyze the chloroplast genome sequence. The results showed that this cultivar chloroplast genome is 121 883 bp in length with only one copy of the IR region. It encodes 109 genes, including 4 ribosomal RNA (rRNA) genes, 29 transfer RNA (tRNA) genes, 75 protein-coding genes, and 1 pseudogene. Compared with other legume species, V. sativa chloroplast genes have few differences in size, order, and structure. This genome sequence has been registered in GenBank under accession number KU053796. The successful sequencing of this genome contributes to chloroplast molecular biology and will be useful in studying genetic breeding and molecular evolution of the common vetch.
箭筈豌豆(Vicia sativa)是自花授粉二倍体一年生豆科牧草。通常用作干草或青贮饲料, 是一种优质牧草, 也可作为高产肥田的优质绿肥[1]。“ 兰箭3号” 春箭筈豌豆是由南志标等[2]选育完成牧草新品种, 并于2011年通过了全国草品种审定委员会的审定。其适宜种植在以青藏高原为主体的草原牧区, 具有早熟、抗寒性强、生育期短等优良特性, 为我国高海拔地区的反刍动物提供优质的蛋白粗饲料[2, 3]。
叶绿体是绿色植物的光合作用中心, 而其中的叶绿素可以吸收光能, 并将光能转化成ATP和NADPH, 同时释放氧气。除了光合作用, 叶绿体在植物细胞中的许多生化过程中均具有重要功能, 如叶绿体氮代谢、硫酸盐还原以及脂质、氨基酸、维生素、功能淀粉等的合成[4, 5]。作为半自主性细胞器, 叶绿体具有自身的DNA以及蛋白质合成体系, 属母系遗传[6]。
叶绿体基因组独立于核基因组, 具有单独的转录和转运系统[7]。研究者通过测序和基因定位, 证实植物的质体(叶绿体和线粒体)基因组一般是非常保守的, 且大多数绿色植物叶绿体基因组是一个四分体结构, 分为一个小的单拷贝片段(small single copy, SSC)、一个大的单拷贝片段(large single copy, LSC)以及一对编码相同, 方向相反的序列IRa和IRb(inverted reapet sequence, IR)[8]。也有少数豆科植物的叶绿体基因组结构有所不同, 如温带豆科植物三叶草(Trifolium subterraneum)、蒺藜苜蓿(Medicago truncatula)、鹰嘴豆(Cicer arietinum)等因一个反向重复序列完全丢失而具有特殊的叶绿体基因组结构[9], 从而呈现出一个三分体结构。而热带豆科植物如大豆(Glycine max)、百脉根(Lotus corniculatus)则具有完整的一对反向重复序列[10, 11]。植物叶绿体全基因组大小通常为120~170 kb, 包括120~130个基因, 且其基因容量和基因排列顺序非常保守[11, 12]。绿色植物叶绿体基因组编码的基因主要分为三大类:一是与自我复制有关的基因, 如转运RNA(tRNA)、核糖体RNA(rRNA)和叶绿体RNA聚合酶合成有关的3个亚基; 二是与光合作用(光合过程)有关的基因; 三是NADH脱氢酶基因、开放阅读框(ORF)以及带有内含子读码结构(IRF)基因[13]。
对于复杂庞大的核基因组而言, 完成其全基因组测序耗时、费力而且成本高, 而仅仅比较个别基因的遗传变异又无法准确分析物种的地理起源、系统发生关系等问题[14]。近几年的研究表明, 叶绿体基因组遗传转化比核转化具有明显的优势, 叶绿体基因具有片段较小(120~170 kb)[11], 测序成本低的优点, 叶绿体其基因组的结构序列信息和序列信息结构在研究物种起源、进化、演变及不同物种之间的亲缘关系等方面具有很好的效果[15]。据统计, 截至2015年2月, NCBI(美国国家生物技术中心)数据库中已经收集了579条植物叶绿体基因组全序列[16]。因植物光合作用严格受遗传控制, 所以了解叶绿体基因组的基因功能, 分析叶绿体的系统发生关系及作用, 对提高作物光合效率、深入研究改良作物特性、探索细胞器的起源和进化具有重要意义[17]。“ 兰箭3号” 叶绿体基因组的测序成功, 可有效促进箭筈豌豆遗传育种和分子进化研究, 对牧草资源的开发利用具有很大的价值。
试验所用“ 兰箭3号” 春箭筈豌豆种子由兰州大学草地农业科技学院提供, 通过种子萌发, 长出复叶后采集新鲜幼嫩叶片, 4 ℃暗处理48 h后提取叶绿体DNA。
1.2.1 叶绿体DNA的提取与纯化 “ 兰箭3号” 叶绿体DNA的提取与纯化采用DNase I法[18], 主要包含5个步骤:叶片的预处理, 叶绿体的初步分离, 叶绿体的纯化, 叶绿体的裂解和叶绿体DNA的纯化。
1.2.2 测序及序列拼接 经纯化后的“ 兰箭3号” 叶绿体DNA样品送由华大科技采用全基因组鸟枪法测序策略以及Illumina Hiseq 2000测序系统进行测序。即通过利用单分子阵列实现在小型芯片(Flow Cell)上进行桥式PCR反应[19]。叶绿体基因组全序列的组装用SOAP de novo软件[20]来完成。通过GenomeVx在线软件(http://wolfe.ucd.ie/GenomeVx/)获得“ 兰箭3号” 的叶绿体基因组序列物理图谱。
1.2.3 基因组序列注释和提交 叶绿体全基因组注释采用NCBI指定的软件Sequin[21](下载页面:http://www.ncbi.nlm.nih.gov/Sequin/index.html), 序列提交通过向NCBI发送邮件(E-mail:gb-admin@ncbi.nlm.nih.gov), 把利用Sequin软件注释好的序列数据上传, 或在NCBI在线提交(在线提交页面:http://www.ncbi.nlm.nih.gov/BankIt/)。
1.2.4 基因组序列进化分析 叶绿体基因组的进化分析及进化树的构建采用MEGA软件[22], 利用软件中的最大简约法(MP)和最大似然法(ML)对数据进行分析。
“ 兰箭3号” 叶绿体全基因组测序得到1 649.25 Mb的原始数据, 测序深度为10 995X, 其中包含17 293 639个片段(reads), 其标准片段长度为100 bp, 通过去除低质量序列片段(质量值小于等于7)、有接头污染的片段、有小插入的片段以及片段1和片段2分别完全一样的片段后[23, 24], 得到15 659 434个片段参与组装, 组装后形成71个有效叠连群, 其中最大叠连群为13 780 bp, N50(覆盖所有核苷酸50%的最大序列重叠群长度)为125 252 bp, 采用过滤后全部的数据做组装分析, 根据总长和N50挑选组装最好的版本, 最终获得GC含量为35.2%的环形叶绿体基因组。
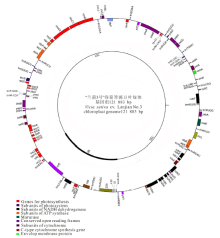
“ 兰箭3号” 叶绿体基因组大小为121 883 bp, 采用Sequin软件注释基因109个, 并通过GenomeVx在线软件(http://wolfe.ucd.ie/GenomeVx/)获得其叶绿体基因组序列物理图谱(图1)。和其它物种相似, “ 兰箭3号” 叶绿体基因组编码的基因主要分为三大类:一是与自我复制有关的基因57个, 包括rRNA基因4个、tRNA基因29个以及编码叶绿体RNA聚合酶的3个亚基(大亚基、小亚基和依赖DNA的RNA聚合酶)基因; 二是与光合作用有关的基因45个; 三是ORF和其它编码蛋白的基因4个以及未知功能基因3个。这三大类基因可分为17个小类, 与光合作用有关的基因种类最多, 可分为7类(表1)。
| 表1 “ 兰箭3号” 春箭筈豌豆叶绿体基因组注释基因列表 Table 1 List of genes found in V. sativa cv. Lanjian No.3 chloroplast genome |
与大部分高等植物叶绿体基因组高度保守的四分体结构相比[8], “ 兰箭3号” 叶绿体基因组在系统进化、发育过程中多次发生重组, 造成一个反向重复序列完全丢失, 因而具有特殊的叶绿体基因组结构。其结构包括一个反向重复序列IR、一个大的单拷贝区域和一个小的单拷贝区域。除此之外, “ 兰箭3号” 叶绿体基因组中rbcL和rps16基因之间出现了一个单一的大约50 kb倒置, 类似于原始被子植物基因顺序。同时, 在“ 兰箭3号” 中也发现了rpl22, ycf4, infA基因的缺失, clpP和rps12基因内含子的缺失以及RNA编辑现象。
“ 兰箭3号” 叶绿体基因组中, 共有16个基因包含1个或两个内含子, 其中11个基因是蛋白质编码基因, 另外5个是tRNA基因。16个含内含子的基因中, trnK-UUU具有最大的内含子(2 443 bp), 而matK基因位于其中。trnL-UAA基因具有最小的内含子(273 bp)。ycf3基因是唯一一个具有两个内含子的基因, 分别为740和782 bp(表2)。
| 表2 “ 兰箭3号” 叶绿体基因组中含有内含子的基因及其内含子和外显子的长度 Table 2 Genes with introns in the Lanjian No.3 chloroplast genome and lengths of exons and introns |
该75个蛋白编码基因共编码20 444个密码子, 在这些密码子中, 1 972个(9.65%)编码异亮氨酸, 235个(1.15%)编码谷氨酰胺, 这分别是“ 兰箭3号” 叶绿体基因组中编码最多(最普遍)和最少的氨基酸, 并且3个终止密码子中UGA在“ 兰箭3号” 叶绿体基因组中使用最频繁(UGA 43.86%, UAG 36.67%和UAA 19.47%), 其它密码子的使用见表3。通过对“ 兰箭3号” 叶绿体基因组中蛋白编码基因和tRNA基因共104个基因序列分析, 得出其相对同义密码子的使用(RSCU)(表4)。RSCU> 1表示该密码子偏好, RSCU< 1表示该密码子使用率较低, RSCU=1表示该密码子没有偏好性。
| 表3 “ 兰箭3号” 叶绿体基因组密码子使用及反密码子的鉴定 Table 3 Codon usage and codon-anticodon recognition pattern for tRNA in the V. sativa cv. Lanjian No.3 cp genome |
| 表4 几种叶绿体基因组基本特征比较 Table 4 Comparison of general features of some plastid genomes |
本研究比较了“ 兰箭3号” 、蚕豆(V. faba)、豌豆(Pisum sativum)、扁豆(Lens culinaris)、山黧豆(Lathyrus venosus)、蒺藜苜蓿以及大豆共7个豆科植物的叶绿体基因种类、排列顺序及结构大小。经过分析发现, 除大豆外的其余几个豆科植物叶绿体基因在种类和数目上有极少数不一致, 表现十分保守(表5), 表现为叶绿体基因组全长120 kb左右, 编码蛋白基因75个、rRNA 4个以及tRNA 29个左右。大豆叶绿体基因组全长比其余6个豆科植物高出近30 kb, 编码蛋白基因128个、rRNA 8个以及tRNA 37个[24]。7个豆科植物中, 蚕豆GC含量最高, 为38.9%。
本研究选取7个豆科植物叶绿体基因组的3个常见叶绿体基因(rpoA, rbcL和ndhK), 并以拟南芥(Arabidopsis thaliana)作为外类群(outgroup), 对“ 兰箭3号” 进行进化分析, 这些基因序列均来自NCBI数据库。MP分析(500次重复)产生一个最简约系统树, 一致性指数(consistency index)为0.810, 保留指数(retention index)为0.887, 综合指数(composite index)为0.780。ML分析(500次重复)产生最大似然系统树[-lnL(unconstrained)](图2), 发现“ 兰箭3号” 与蚕豆亲缘关系较近, 自展支持率超过了70%, 均属于野豌豆属的植物, 而大豆与其亲缘关系最远, 3个基因分析其进化趋势一致。并且和预期一样, 作为外类群的拟南芥, 聚在每个基因的最外侧。ML分析拓扑结构与MP分析相同。
 | 图2 不同豆科植物叶绿体基因组的ML系统进化树 注:图中仅显示大于50%的自展支持率, 基因序列来自NCBI数据库, 拟南芥作为外类群(序列号见表4)。Fig.2 ML phylogenetic tree in 3 genes (rpoA, rbcL and ndhK) of different plants in the legume family Note:Bootstrap support values(> 50%)are indicated above the branches with Arabidopsis thaliana as outgroup. Sequence data was obtained from NCBI with Genbank accession numbers provided in Table 4. |
叶绿体是光合作用的主要场所, 独立于植物细胞核具有高度保守的基因组结构, 且独立遗传[25]。自1986年第一次获得烟草(Nicotiana tabacum)叶绿体基因组全序列以来[26], 叶绿体基因组研究在植物光合作用机理、叶绿体基因功能、物种起源及系统进化等方面广泛应用。
前人研究证明, 大部分被子植物叶绿体基因组结构为高度保守的四分体结构[8], 也有少数温带豆科植物的叶绿体基因组结构有所不同, 如三叶草[27]、蒺藜苜蓿[28]、鹰嘴豆[29]等因一个反向重复序列完全丢失而具有特殊的叶绿体基因组结构(LSC, SSC和IR), 热带豆科植物如大豆[25]、百脉根[30]则具有完整的一对反向重复序列(LSC, SSC, IRa和IRb)。本研究中“ 兰箭3号” 叶绿体基因组结构与上述温带豆科植物叶绿体基因组研究结果一致。
与其它高等植物相比, 部分豆科植物叶绿体基因组在进化过程中多次发生重组[29], 从而造成一系列结构变化, 除一个拷贝的IR区域缺失, 还包括rpl23、rpl22、accD、ycf1、ycf2、ycf4、rps16和infA等基因部分甚至全部从豆科植物叶绿体基因组中缺失及clpP和rps12基因内含子的缺失, 有的甚至出现了RNA编辑现象[31]。本研究中“ 兰箭3号” 叶绿体全基因组中发现的rpl22, ycf4, infA基因的缺失, clpP和rps12基因内含子的缺失以及RNA编辑现象的结果与前人研究相符[29]。
在大部分生物中, 密码子的使用频率并不相等, 而通过长期的进化, 会形成某一物种或某一基因对特定的同义密码子的使用偏好, 这种现象称为密码子偏好性[32]。研究表明:不同物种甚至同一物种的不同基因具有特异的密码子使用偏好, 而造成这种特异性的隐藏因素就是物种与基因在长期进化过程中选择、突变和漂变的综合作用[33]。并且密码子的偏好性分析对宿主中外源基因表达水平的提高具有重要的意义[32]。通过比较分析, 发现“ 兰箭3号” 与其近缘种蚕豆在其最优密码子的使用上很相似, 但也存在差异。这与前人研究结果相符[33]。
由于叶绿体基因组结构简单、相对保守且属于母系遗传, 所以叶绿体简单重复序列是一种高效的分子标记, 叶绿体基因组分子标记(cpSSRs)广泛应用于杂交育种、生物地理学和群体遗传学的研究[34]。在“ 兰箭3号” 中, 我们发现了3种cpSSRs类型, 包括单碱基重复、二碱基重复和三碱基重复。这为以后箭筈豌豆物种鉴定以及群体和个体水平的遗传差异分析奠定基础。
研究中使用一个基因研究进化关系, 很难准确判断他们的亲缘关系[35], 而本研究采用叶绿体基因组3个蛋白编码基因进行进化关系分析, 提高了研究其进化关系的准确性, 并且3个基因分析进化关系一致。由ML进化树看出, 大豆与野豌豆属植物亲缘关系较远。这是因为大豆[25]叶绿体基因组是四分体结构, 而“ 兰箭3号” 和蚕豆[36]均为特殊的叶绿体基因组结构, 这与传统分类学结果一致, 也验证了特殊结构的叶绿体基因组在进化过程中发生了重组[29]。
目前, 植物遗传转化体系多以核基因转化为基础, 随生物技术不断发展, 人们发现核基因组转化面临外源基因难以控制、核基因组基因数目庞大, 基因功能和遗传背景较复杂等难以克服的缺陷[37]。而叶绿体是半自主细胞器, 具有独立遗传体系, 可防止外源基因污染、基因泄漏等问题发生[37]。所以近年来, 叶绿体基因组遗传转化技术快速发展, 研究证实植物叶绿体基因组中编码的目的蛋白占可溶性蛋白的46.1%, 很适合作为遗传转化的载体[38, 39], 因此了解叶绿体的基因功能和序列信息是叶绿体转化的基础[14]。本研究采用高通量测序技术成功对“ 兰箭3号” 叶绿体全基因组进行了测序, 获得了其全长序列并对109个基因进行了注释, 这为箭筈豌豆的遗传育种和其它豆科植物叶绿体基因组的研究奠定了基础。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|